| 登録情報 | データベース: PDB / ID: 8tzr
|
|---|
| タイトル | Structure of human Wnt3a bound to WLS and CALR |
|---|
 要素 要素 | - Calreticulin
- Protein Wnt-3a
- Protein wntless homolog
|
|---|
 キーワード キーワード | SIGNALING PROTEIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Wnt signaling pathway involved in forebrain neuroblast division / positive regulation of dermatome development / axis elongation involved in somitogenesis / calcium ion transmembrane transport via low voltage-gated calcium channel / : / positive regulation of collateral sprouting in absence of injury / positive regulation of mesodermal cell fate specification / paraxial mesodermal cell fate commitment / cell proliferation in midbrain / spinal cord association neuron differentiation ...Wnt signaling pathway involved in forebrain neuroblast division / positive regulation of dermatome development / axis elongation involved in somitogenesis / calcium ion transmembrane transport via low voltage-gated calcium channel / : / positive regulation of collateral sprouting in absence of injury / positive regulation of mesodermal cell fate specification / paraxial mesodermal cell fate commitment / cell proliferation in midbrain / spinal cord association neuron differentiation / Formation of the posterior neural plate / Wnt protein secretion / Calnexin/calreticulin cycle / COP9 signalosome assembly / positive regulation of cell-cell adhesion mediated by cadherin / cytolytic granule / Wnt-Frizzled-LRP5/6 complex / positive regulation of Wnt protein secretion / Negative regulation of TCF-dependent signaling by WNT ligand antagonists / synaptic vesicle recycling / Signaling by RNF43 mutants / positive regulation of dendritic cell chemotaxis / WNT ligand biogenesis and trafficking / positive regulation of cardiac muscle cell differentiation / cortical granule / Assembly of Viral Components at the Budding Site / negative regulation of trophoblast cell migration / ATF6 (ATF6-alpha) activates chaperone genes / cell proliferation in forebrain / negative regulation of retinoic acid receptor signaling pathway / cellular response to electrical stimulus / endoplasmic reticulum quality control compartment / nuclear receptor-mediated glucocorticoid signaling pathway / regulation of meiotic nuclear division / secondary palate development / complement component C1q complex binding / Specification of the neural plate border / negative regulation of axon extension involved in axon guidance / sequestering of calcium ion / cementum mineralization / response to glycoside / somatic stem cell division / non-canonical Wnt signaling pathway / co-receptor binding / sarcoplasmic reticulum lumen / cardiac muscle cell fate commitment / presynapse assembly / protein folding in endoplasmic reticulum / hindbrain development / positive regulation of skeletal muscle tissue development / nuclear export signal receptor activity / dorsal/ventral neural tube patterning / negative regulation of dopaminergic neuron differentiation / negative regulation of intracellular steroid hormone receptor signaling pathway / hormone binding / midbrain dopaminergic neuron differentiation / Wnt-protein binding / regulation of postsynapse to nucleus signaling pathway / cardiac muscle cell differentiation / molecular sequestering activity / exocrine pancreas development / mammary gland development / post-anal tail morphogenesis / positive regulation of neural precursor cell proliferation / frizzled binding / positive regulation of hepatocyte proliferation / Class B/2 (Secretin family receptors) / Disassembly of the destruction complex and recruitment of AXIN to the membrane / myoblast differentiation / anterior/posterior axis specification / protein maturation by protein folding / Scavenging by Class F Receptors / Scavenging by Class A Receptors / cortical actin cytoskeleton organization / midbrain development / nuclear androgen receptor binding / inner ear morphogenesis / regulation of synapse organization / cellular response to lithium ion / response to testosterone / negative regulation of fat cell differentiation / B cell proliferation / fat cell differentiation / heart looping / Formation of paraxial mesoderm / skeletal muscle cell differentiation / positive regulation of receptor internalization / hemopoiesis / regulation of presynapse assembly / mesoderm formation / protein localization to nucleus / negative regulation of neuron differentiation / positive regulation of Wnt signaling pathway / smooth endoplasmic reticulum / canonical Wnt signaling pathway / cell fate commitment / endomembrane system / positive regulation of cell cycle / regulation of microtubule cytoskeleton organization / ERAD pathway類似検索 - 分子機能 Wnt-3 protein / Protein wntless / : / Wntless-like, transmembrane domain / Wnt protein, conserved site / Wnt-1 family signature. / Wnt / Wnt, C-terminal domain / wnt family / found in Wnt-1 ...Wnt-3 protein / Protein wntless / : / Wntless-like, transmembrane domain / Wnt protein, conserved site / Wnt-1 family signature. / Wnt / Wnt, C-terminal domain / wnt family / found in Wnt-1 / Calreticulin / Calreticulin family repeated motif signature. / Calreticulin/calnexin / Calreticulin/calnexin, P domain superfamily / Calreticulin/calnexin, conserved site / Calreticulin family / Calreticulin family signature 1. / Calreticulin family signature 2. / Endoplasmic reticulum targeting sequence. / Concanavalin A-like lectin/glucanase domain superfamily類似検索 - ドメイン・相同性 PALMITOLEIC ACID / Chem-POV / Calreticulin / Protein Wnt-3a / Protein wntless homolog類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å |
|---|
 データ登録者 データ登録者 | Qi, X. / Hu, Q. / Li, X. |
|---|
| 資金援助 |  米国, 3件 米国, 3件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM135343 |  米国 米国 | | National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | HL160487 |  米国 米国 | | Welch Foundation | I-1957 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Structure of human Wnt3a bound to WLS and CALR
著者: Qi, X. / Hu, Q. / Li, X. |
|---|
| 履歴 | | 登録 | 2023年8月27日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2023年10月18日 | Provider: repository / タイプ: Initial release |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8tzr.cif.gz
8tzr.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8tzr.ent.gz
pdb8tzr.ent.gz PDB形式
PDB形式 8tzr.json.gz
8tzr.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8tzr_validation.pdf.gz
8tzr_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8tzr_full_validation.pdf.gz
8tzr_full_validation.pdf.gz 8tzr_validation.xml.gz
8tzr_validation.xml.gz 8tzr_validation.cif.gz
8tzr_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/tz/8tzr
https://data.pdbj.org/pub/pdb/validation_reports/tz/8tzr ftp://data.pdbj.org/pub/pdb/validation_reports/tz/8tzr
ftp://data.pdbj.org/pub/pdb/validation_reports/tz/8tzr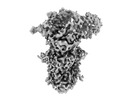
 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: WNT3A / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: WNT3A / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P56704
Homo sapiens (ヒト) / 参照: UniProt: P56704 Homo sapiens (ヒト) / 遺伝子: WLS / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: WLS / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q5T9L3
Homo sapiens (ヒト) / 参照: UniProt: Q5T9L3 Homo sapiens (ヒト) / 参照: UniProt: P27797
Homo sapiens (ヒト) / 参照: UniProt: P27797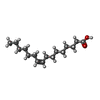


 試料調製
試料調製 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) 電子顕微鏡撮影
電子顕微鏡撮影
 FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM
FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM 解析
解析 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj









