+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8rhn | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the 55LCC ATPase complex | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | DNA BINDING PROTEIN / DNA replication / AAA+ ATPases / proteostasis | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報preribosome binding / non-chaperonin molecular chaperone ATPase / ribosomal large subunit biogenesis / brain development / spindle / DNA replication / spermatogenesis / cell differentiation / cell division / DNA repair ...preribosome binding / non-chaperonin molecular chaperone ATPase / ribosomal large subunit biogenesis / brain development / spindle / DNA replication / spermatogenesis / cell differentiation / cell division / DNA repair / ATP hydrolysis activity / mitochondrion / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | ||||||||||||
 データ登録者 データ登録者 | Foglizzo, M. / Degtjarik, O. / Zeqiraj, E. | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: The SPATA5-SPATA5L1 ATPase complex directs replisome proteostasis to ensure genome integrity. 著者: Vidhya Krishnamoorthy / Martina Foglizzo / Robert L Dilley / Angela Wu / Arindam Datta / Parul Dutta / Lisa J Campbell / Oksana Degtjarik / Laura J Musgrove / Antonio N Calabrese / Elton ...著者: Vidhya Krishnamoorthy / Martina Foglizzo / Robert L Dilley / Angela Wu / Arindam Datta / Parul Dutta / Lisa J Campbell / Oksana Degtjarik / Laura J Musgrove / Antonio N Calabrese / Elton Zeqiraj / Roger A Greenberg /   要旨: Ubiquitin-dependent unfolding of the CMG helicase by VCP/p97 is required to terminate DNA replication. Other replisome components are not processed in the same fashion, suggesting that additional ...Ubiquitin-dependent unfolding of the CMG helicase by VCP/p97 is required to terminate DNA replication. Other replisome components are not processed in the same fashion, suggesting that additional mechanisms underlie replication protein turnover. Here, we identify replisome factor interactions with a protein complex composed of AAA+ ATPases SPATA5-SPATA5L1 together with heterodimeric partners C1orf109-CINP (55LCC). An integrative structural biology approach revealed a molecular architecture of SPATA5-SPATA5L1 N-terminal domains interacting with C1orf109-CINP to form a funnel-like structure above a cylindrically shaped ATPase motor. Deficiency in the 55LCC complex elicited ubiquitin-independent proteotoxicity, replication stress, and severe chromosome instability. 55LCC showed ATPase activity that was specifically enhanced by replication fork DNA and was coupled to cysteine protease-dependent cleavage of replisome substrates in response to replication fork damage. These findings define 55LCC-mediated proteostasis as critical for replication fork progression and genome stability and provide a rationale for pathogenic variants seen in associated human neurodevelopmental disorders. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8rhn.cif.gz 8rhn.cif.gz | 950.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8rhn.ent.gz pdb8rhn.ent.gz | 724.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8rhn.json.gz 8rhn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rh/8rhn https://data.pdbj.org/pub/pdb/validation_reports/rh/8rhn ftp://data.pdbj.org/pub/pdb/validation_reports/rh/8rhn ftp://data.pdbj.org/pub/pdb/validation_reports/rh/8rhn | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | 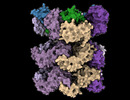 19177MC  8cihC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 101307.883 Da / 分子数: 8 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: AFG2A, SPAF, SPATA5 Homo sapiens (ヒト) / 遺伝子: AFG2A, SPAF, SPATA5発現宿主:  参照: UniProt: Q8NB90, non-chaperonin molecular chaperone ATPase #2: タンパク質 | 分子量: 83362.836 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: AFG2B, SPATA5L1 Homo sapiens (ヒト) / 遺伝子: AFG2B, SPATA5L1発現宿主:  参照: UniProt: Q9BVQ7, non-chaperonin molecular chaperone ATPase #3: タンパク質 | 分子量: 29439.279 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) Homo sapiens (ヒト)発現宿主:  参照: UniProt: B4DRQ5 #4: タンパク質 | 分子量: 27462.139 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CINP Homo sapiens (ヒト) / 遺伝子: CINP発現宿主:  参照: UniProt: Q9BW66 |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Structure of the 55LCC ATPase complex / タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT | |||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.649 MDa / 実験値: NO | |||||||||||||||||||||||||||||||||||
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:  | |||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 詳細: 25 mM Tris pH 7.5, 400 mM NaCl, 5% (v/v) glycerol, 1 mM EGTA, 2 mM ADP, 2 mM MgCl2 and 1 mM TCEP | |||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| |||||||||||||||||||||||||||||||||||
| 試料 | 濃度: 3.7 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||||||||||||||||||||||
| 試料支持 | 詳細: Quantifoil R3.5/1 200-mesh grids (Quantifoil Micro Tools GmbH) were glow-discharged for 30 s at 12 mA and 0.38 mBar pressure using a PELCO easiGlow system (Ted Pella). グリッドの材料: COPPER / グリッドのサイズ: 200 divisions/in. / グリッドのタイプ: Quantifoil R3.5/1 | |||||||||||||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K / 詳細: blot force = 0 N blot time = 8 s |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 165000 X / 最大 デフォーカス(公称値): 3100 nm / 最小 デフォーカス(公称値): 1200 nm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 2.99 sec. / 電子線照射量: 37.6 e/Å2 フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) 撮影したグリッド数: 1 / 実像数: 27595 |
| 電子光学装置 | エネルギーフィルター名称: TFS Selectris / エネルギーフィルタースリット幅: 10 eV / 位相板: VOLTA PHASE PLATE |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 4182380 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C2 (2回回転対称) | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 4.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 165778 / クラス平均像の数: 2 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 127.55 / プロトコル: RIGID BODY FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | Source name: AlphaFold / タイプ: in silico model | ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj



