+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8p7c | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of METTL6 tRNA SerRS complex in a 2:2:2 stoichiometry | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | RNA BINDING PROTEIN / METTL6 / tRNA / SerRS / Serine tRNA / 3-Methylcytosine | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報selenocysteine-tRNA ligase activity / tRNA (cytidine-3-)-methyltransferase activity / negative regulation of vascular endothelial growth factor production / selenocysteine incorporation / serine-tRNA ligase / serine-tRNA ligase activity / seryl-tRNA aminoacylation / Cytosolic tRNA aminoacylation / tRNA modification / tRNA methylation ...selenocysteine-tRNA ligase activity / tRNA (cytidine-3-)-methyltransferase activity / negative regulation of vascular endothelial growth factor production / selenocysteine incorporation / serine-tRNA ligase / serine-tRNA ligase activity / seryl-tRNA aminoacylation / Cytosolic tRNA aminoacylation / tRNA modification / tRNA methylation / Selenocysteine synthesis / negative regulation of angiogenesis / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / molecular adaptor activity / cytoplasmic translation / tRNA binding / translation / RNA polymerase II cis-regulatory region sequence-specific DNA binding / enzyme binding / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / extracellular exosome / ATP binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Throll, P. / Dolce, L.G. / Kowalinski, E. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 資金援助 | 1件
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural basis of tRNA recognition by the mC RNA methyltransferase METTL6 in complex with SerRS seryl-tRNA synthetase. 著者: Philipp Throll / Luciano G Dolce / Palma Rico-Lastres / Katharina Arnold / Laura Tengo / Shibom Basu / Stefanie Kaiser / Robert Schneider / Eva Kowalinski /   要旨: Methylation of cytosine 32 in the anticodon loop of tRNAs to 3-methylcytosine (mC) is crucial for cellular translation fidelity. Misregulation of the RNA methyltransferases setting this modification ...Methylation of cytosine 32 in the anticodon loop of tRNAs to 3-methylcytosine (mC) is crucial for cellular translation fidelity. Misregulation of the RNA methyltransferases setting this modification can cause aggressive cancers and metabolic disturbances. Here, we report the cryo-electron microscopy structure of the human mC tRNA methyltransferase METTL6 in complex with seryl-tRNA synthetase (SerRS) and their common substrate tRNA. Through the complex structure, we identify the tRNA-binding domain of METTL6. We show that SerRS acts as the tRNA substrate selection factor for METTL6. We demonstrate that SerRS augments the methylation activity of METTL6 and that direct contacts between METTL6 and SerRS are necessary for efficient tRNA methylation. Finally, on the basis of the structure of METTL6 in complex with SerRS and tRNA, we postulate a universal tRNA-binding mode for mC RNA methyltransferases, including METTL2 and METTL8, suggesting that these mammalian paralogs use similar ways to engage their respective tRNA substrates and cofactors. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8p7c.cif.gz 8p7c.cif.gz | 339.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8p7c.ent.gz pdb8p7c.ent.gz | 264.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8p7c.json.gz 8p7c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8p7c_validation.pdf.gz 8p7c_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8p7c_full_validation.pdf.gz 8p7c_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  8p7c_validation.xml.gz 8p7c_validation.xml.gz | 76.6 KB | 表示 | |
| CIF形式データ |  8p7c_validation.cif.gz 8p7c_validation.cif.gz | 109.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/p7/8p7c https://data.pdbj.org/pub/pdb/validation_reports/p7/8p7c ftp://data.pdbj.org/pub/pdb/validation_reports/p7/8p7c ftp://data.pdbj.org/pub/pdb/validation_reports/p7/8p7c | HTTPS FTP |
-関連構造データ
| 関連構造データ |  17529MC  8owxC 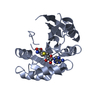 8owyC  8p7bC  8p7dC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 2種, 4分子 BDAC
| #1: タンパク質 | 分子量: 58863.211 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: SARS1, SARS, SERS / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SARS1, SARS, SERS / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P49591, serine-tRNA ligase Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P49591, serine-tRNA ligase#2: タンパク質 | 分子量: 33296.055 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: METTL6 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: METTL6 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ)参照: UniProt: Q8TCB7, 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの |
|---|
-RNA鎖 , 1種, 2分子 RT
| #3: RNA鎖 | 分子量: 27620.635 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
|---|
-非ポリマー , 3種, 10分子 




| #4: 化合物 | | #5: 化合物 | ChemComp-MG / #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | N |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: METTL6 tRNA SerRS complex in a 2:2:2 stoichiometry / タイプ: COMPLEX / Entity ID: #1-#3 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1800 nm / 最小 デフォーカス(公称値): 800 nm |
| 撮影 | 電子線照射量: 63.27 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 対称性 | 点対称性: C2 (2回回転対称) | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 105928 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj

































