[English] 日本語
 Yorodumi
Yorodumi- PDB-8fgv: Structure of rat neuronal nitric oxide synthase H692F mutant heme... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 8fgv | ||||||
|---|---|---|---|---|---|---|---|
| Title | Structure of rat neuronal nitric oxide synthase H692F mutant heme domain in complex with 6-(5-(2-(dimethylamino)ethyl)-2,3-difluorophenethyl)-4-methoxypyridin-2-amine | ||||||
 Components Components | Nitric oxide synthase, brain | ||||||
 Keywords Keywords | OXIDOREDUCTASE/OXIDOREDUCTASE INHIBITOR / nitric oxide synthase inhibitor / heme enzyme / OXIDOREDUCTASE / OXIDOREDUCTASE-Inhibitor complex / OXIDOREDUCTASE-OXIDOREDUCTASE INHIBITOR complex | ||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of hepatic stellate cell contraction / Nitric oxide stimulates guanylate cyclase / positive regulation of adenylate cyclase-activating adrenergic receptor signaling pathway / negative regulation of iron ion transmembrane transport / response to vitamin B3 / postsynaptic specialization, intracellular component / ROS and RNS production in phagocytes / azurophil granule / Ion homeostasis / synaptic signaling by nitric oxide ...negative regulation of hepatic stellate cell contraction / Nitric oxide stimulates guanylate cyclase / positive regulation of adenylate cyclase-activating adrenergic receptor signaling pathway / negative regulation of iron ion transmembrane transport / response to vitamin B3 / postsynaptic specialization, intracellular component / ROS and RNS production in phagocytes / azurophil granule / Ion homeostasis / synaptic signaling by nitric oxide / negative regulation of vasoconstriction / response to nitric oxide / positive regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / response to vitamin E / negative regulation of cytosolic calcium ion concentration / positive regulation of sodium ion transmembrane transport / peptidyl-cysteine S-nitrosylase activity / positive regulation of the force of heart contraction / cadmium ion binding / negative regulation of calcium ion transport / negative regulation of potassium ion transport / regulation of postsynaptic membrane potential / nitric oxide mediated signal transduction / sodium channel regulator activity / nitric-oxide synthase (NADPH) / regulation of neurogenesis / negative regulation of serotonin uptake / nitric-oxide synthase activity / xenobiotic catabolic process / multicellular organismal response to stress / L-arginine catabolic process / NADPH binding / postsynaptic density, intracellular component / striated muscle contraction / nitric oxide-cGMP-mediated signaling / negative regulation of blood pressure / regulation of sodium ion transport / response to hormone / behavioral response to cocaine / nitric oxide metabolic process / nitric oxide biosynthetic process / photoreceptor inner segment / cellular response to epinephrine stimulus / T-tubule / sarcoplasmic reticulum membrane / secretory granule / calyx of Held / sarcoplasmic reticulum / positive regulation of long-term synaptic potentiation / response to activity / cell periphery / response to nicotine / response to nutrient levels / phosphoprotein binding / establishment of protein localization / establishment of localization in cell / female pregnancy / cellular response to mechanical stimulus / negative regulation of insulin secretion / sarcolemma / response to peptide hormone / caveola / potassium ion transport / response to lead ion / cellular response to growth factor stimulus / response to estrogen / vasodilation / Z disc / calcium-dependent protein binding / calcium ion transport / FMN binding / NADP binding / flavin adenine dinucleotide binding / positive regulation of neuron apoptotic process / ATPase binding / response to heat / scaffold protein binding / response to ethanol / nuclear membrane / response to lipopolysaccharide / dendritic spine / negative regulation of neuron apoptotic process / RNA polymerase II-specific DNA-binding transcription factor binding / transmembrane transporter binding / response to hypoxia / mitochondrial outer membrane / cytoskeleton / calmodulin binding / postsynaptic density / membrane raft / negative regulation of cell population proliferation / heme binding / synapse / dendrite / negative regulation of apoptotic process / positive regulation of DNA-templated transcription / perinuclear region of cytoplasm / glutamatergic synapse / enzyme binding / positive regulation of transcription by RNA polymerase II Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  FOURIER SYNTHESIS / Resolution: 1.848 Å FOURIER SYNTHESIS / Resolution: 1.848 Å | ||||||
 Authors Authors | Li, H. / Poulos, T.L. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: J.Med.Chem. / Year: 2023 Journal: J.Med.Chem. / Year: 2023Title: Potent, Selective, and Membrane Permeable 2-Amino-4-Substituted Pyridine-Based Neuronal Nitric Oxide Synthase Inhibitors. Authors: Vasu, D. / Do, H.T. / Li, H. / Hardy, C.D. / Awasthi, A. / Poulos, T.L. / Silverman, R.B. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  8fgv.cif.gz 8fgv.cif.gz | 365.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb8fgv.ent.gz pdb8fgv.ent.gz | 296.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  8fgv.json.gz 8fgv.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  8fgv_validation.pdf.gz 8fgv_validation.pdf.gz | 1.7 MB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  8fgv_full_validation.pdf.gz 8fgv_full_validation.pdf.gz | 1.8 MB | Display | |
| Data in XML |  8fgv_validation.xml.gz 8fgv_validation.xml.gz | 36.7 KB | Display | |
| Data in CIF |  8fgv_validation.cif.gz 8fgv_validation.cif.gz | 50.8 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/fg/8fgv https://data.pdbj.org/pub/pdb/validation_reports/fg/8fgv ftp://data.pdbj.org/pub/pdb/validation_reports/fg/8fgv ftp://data.pdbj.org/pub/pdb/validation_reports/fg/8fgv | HTTPS FTP |
-Related structure data
| Related structure data |  8fg9C  8fgaC  8fgbC  8fgcC  8fgdC  8fgeC  8fgfC  8fggC  8fghC  8fgiC  8fgjC  8fgkC  8fglC  8fgmC  8fgnC  8fgoC  8fgpC  8fgqC  8fgrC  8fgsC  8fgtC  8fguC  1om4S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 48821.555 Da / Num. of mol.: 2 / Mutation: H692F Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
|---|
-Non-polymers , 7 types, 330 molecules 
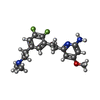











| #2: Chemical | | #3: Chemical | #4: Chemical | #5: Chemical | ChemComp-GOL / | #6: Chemical | #7: Chemical | ChemComp-ZN / | #8: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.48 Å3/Da / Density % sol: 50.4 % / Description: bricks |
|---|---|
| Crystal grow | Temperature: 277 K / Method: vapor diffusion, sitting drop / pH: 5.8 Details: 20-24% PEG3350, 0.1M MES 0.14-0.20M AMMONIUM ACETATE, 10% ETHYLENE GLYCOL, 30uM SDS, 5 mM GSH |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SSRL SSRL  / Beamline: BL12-2 / Wavelength: 0.9795 Å / Beamline: BL12-2 / Wavelength: 0.9795 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Jul 26, 2021 / Details: mirrors |
| Radiation | Monochromator: DOUBLE CRYSTAL SI(III) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9795 Å / Relative weight: 1 |
| Reflection | Resolution: 1.848→39.27 Å / Num. obs: 83747 / % possible obs: 99.6 % / Redundancy: 11.4 % / CC1/2: 0.999 / Rmerge(I) obs: 0.156 / Rpim(I) all: 0.048 / Rrim(I) all: 0.163 / Net I/σ(I): 10.6 |
| Reflection shell | Resolution: 1.85→1.88 Å / Redundancy: 5.8 % / Rmerge(I) obs: 5.331 / Num. unique obs: 4256 / CC1/2: 0.436 / Rpim(I) all: 2.351 / Rrim(I) all: 5.838 / % possible all: 96.8 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  FOURIER SYNTHESIS FOURIER SYNTHESISStarting model: 1OM4 Resolution: 1.848→38.665 Å / SU ML: 0.26 / Cross valid method: THROUGHOUT / σ(F): 0.02 / Phase error: 37.16 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.848→38.665 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller


 PDBj
PDBj









