+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8df0 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Abp1D receptor binding domain | |||||||||
 要素 要素 | Abp1D Receptor Binding Domain | |||||||||
 キーワード キーワード | CELL ADHESION / Chaperone usher pathway adhesin receptor binding domain | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å | |||||||||
 データ登録者 データ登録者 | Tamadonfar, K.O. / Pinkner, J.S. / Dodson, K.W. / Hultgren, S.J. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2023 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2023タイトル: Structure-function correlates of fibrinogen binding by Acinetobacter adhesins critical in catheter-associated urinary tract infections. 著者: Tamadonfar, K.O. / Di Venanzio, G. / Pinkner, J.S. / Dodson, K.W. / Kalas, V. / Zimmerman, M.I. / Bazan Villicana, J. / Bowman, G.R. / Feldman, M.F. / Hultgren, S.J. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8df0.cif.gz 8df0.cif.gz | 191.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8df0.ent.gz pdb8df0.ent.gz | 156.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8df0.json.gz 8df0.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/df/8df0 https://data.pdbj.org/pub/pdb/validation_reports/df/8df0 ftp://data.pdbj.org/pub/pdb/validation_reports/df/8df0 ftp://data.pdbj.org/pub/pdb/validation_reports/df/8df0 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  8dezSC 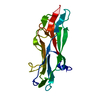 8dkaC S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 18846.119 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア)遺伝子: fimA, AYR68_10060, B7L45_11295, BAA1790NC_1605, BS065_10665, CBL15_10290, CTZ19_10795, D8O08_010130, DLI72_16175, E2532_11475, E2533_08890, E2534_05850, E2535_07595, E2536_00985, E2538_ ...遺伝子: fimA, AYR68_10060, B7L45_11295, BAA1790NC_1605, BS065_10665, CBL15_10290, CTZ19_10795, D8O08_010130, DLI72_16175, E2532_11475, E2533_08890, E2534_05850, E2535_07595, E2536_00985, E2538_06840, E2539_15695, E2540_01165, E2541_05705, EA720_002470, FE003_11010, H1058_10155, HIN86_11385, IAG11_17165, SAMEA104305340_00065 発現宿主:  #2: 水 | ChemComp-HOH / | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.96 Å3/Da / 溶媒含有率: 75.21 % |
|---|---|
| 結晶化 | 温度: 292.15 K / 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: 0.8 M (NH4)2SO4, 0.025 M Citric Acid pH 3.56 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 4.2.2 / 波長: 0.9762 Å / ビームライン: 4.2.2 / 波長: 0.9762 Å |
| 検出器 | タイプ: RDI CMOS_8M / 検出器: CMOS / 日付: 2019年10月25日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9762 Å / 相対比: 1 |
| 反射 | 解像度: 2.1→44.51 Å / Num. obs: 45137 / % possible obs: 99.76 % / 冗長度: 13.9 % / Biso Wilson estimate: 33.97 Å2 / CC1/2: 0.996 / CC star: 0.999 / Rmerge(I) obs: 0.2715 / Rpim(I) all: 0.07507 / Rrim(I) all: 0.2818 / Net I/σ(I): 11.9 |
| 反射 シェル | 解像度: 2.1→2.175 Å / 冗長度: 14 % / Rmerge(I) obs: 2.938 / Num. unique obs: 4370 / CC1/2: 0.434 / CC star: 0.778 / Rpim(I) all: 0.8113 / Rrim(I) all: 3.049 / % possible all: 99.36 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 8DEZ 解像度: 2.1→44.51 Å / SU ML: 0.25 / 交差検証法: THROUGHOUT / σ(F): 1.33 / 位相誤差: 23.8 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 93.76 Å2 / Biso mean: 42.8553 Å2 / Biso min: 23.93 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.1→44.51 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: -5.6049 Å / Origin y: 21.9195 Å / Origin z: -22.4341 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj
