| 登録情報 | データベース: PDB / ID: 8d4v
|
|---|
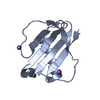
| タイトル | Crystal Structure of Cathepsin G Inhibited by Eap2 from S. aureus |
|---|
 要素 要素 | - Cathepsin G, C-terminal truncated form
- Extracellular Adherence Protein
|
|---|
 キーワード キーワード | HYDROLASE/INHIBITOR / PROTEIN BINDING / Protease Inhibitor / Immune Evasion / Neutrophil / S. aureus / HYDROLASE-INHIBITOR / PROTEIN BINDING complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cathepsin G / biofilm matrix disassembly / neutrophil-mediated killing of gram-positive bacterium / purinergic nucleotide receptor signaling pathway / caspase binding / negative regulation of T cell activation / neutrophil activation / Suppression of apoptosis / Interleukin-1 processing / positive regulation of platelet aggregation ...cathepsin G / biofilm matrix disassembly / neutrophil-mediated killing of gram-positive bacterium / purinergic nucleotide receptor signaling pathway / caspase binding / negative regulation of T cell activation / neutrophil activation / Suppression of apoptosis / Interleukin-1 processing / positive regulation of platelet aggregation / Antimicrobial peptides / Activation of Matrix Metalloproteinases / monocyte chemotaxis / extracellular matrix disassembly / defense response to fungus / Purinergic signaling in leishmaniasis infection / Metabolism of Angiotensinogen to Angiotensins / angiotensin maturation / Degradation of the extracellular matrix / serine-type peptidase activity / secretory granule / protein maturation / cytokine-mediated signaling pathway / positive regulation of immune response / protein processing / platelet activation / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / cytoplasmic stress granule / azurophil granule lumen / antibacterial humoral response / peptidase activity / heparin binding / cellular response to lipopolysaccharide / : / defense response to Gram-negative bacterium / lysosome / defense response to Gram-positive bacterium / protein phosphorylation / immune response / receptor ligand activity / serine-type endopeptidase activity / Neutrophil degranulation / proteolysis / extracellular space / extracellular exosome / extracellular region / nucleus / membrane / plasma membrane / cytosol類似検索 - 分子機能 MAP domain / MAP domain / MAP repeat profile. / Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, serine active site / Serine proteases, trypsin family, histidine active site. / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site. / Serine proteases, trypsin domain profile. / Trypsin-like serine protease ...MAP domain / MAP domain / MAP repeat profile. / Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, serine active site / Serine proteases, trypsin family, histidine active site. / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site. / Serine proteases, trypsin domain profile. / Trypsin-like serine protease / Serine proteases, trypsin domain / Trypsin / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Staphylococcus aureus subsp. aureus Mu50 (黄色ブドウ球菌) Staphylococcus aureus subsp. aureus Mu50 (黄色ブドウ球菌)
 Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.85 Å 分子置換 / 解像度: 1.85 Å |
|---|
 データ登録者 データ登録者 | Gido, C.D. / Herdendorf, T.J. / Geisbrecht, B.V. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | 5R35GM140852 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2023 ジャーナル: J.Biol.Chem. / 年: 2023
タイトル: Characterization of two distinct neutrophil serine protease-binding modes within a Staphylococcus aureus innate immune evasion protein family.
著者: Gido, C.D. / Herdendorf, T.J. / Geisbrecht, B.V. |
|---|
| 履歴 | | 登録 | 2022年6月2日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2022年12月21日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年7月5日 | Group: Database references / カテゴリ: citation
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2023年10月25日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
| 改定 1.3 | 2024年11月13日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Staphylococcus aureus subsp. aureus Mu50 (黄色ブドウ球菌)
Staphylococcus aureus subsp. aureus Mu50 (黄色ブドウ球菌) Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.85 Å
分子置換 / 解像度: 1.85 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: J.Biol.Chem. / 年: 2023
ジャーナル: J.Biol.Chem. / 年: 2023 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8d4v.cif.gz
8d4v.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8d4v.ent.gz
pdb8d4v.ent.gz PDB形式
PDB形式 8d4v.json.gz
8d4v.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8d4v_validation.pdf.gz
8d4v_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8d4v_full_validation.pdf.gz
8d4v_full_validation.pdf.gz 8d4v_validation.xml.gz
8d4v_validation.xml.gz 8d4v_validation.cif.gz
8d4v_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/d4/8d4v
https://data.pdbj.org/pub/pdb/validation_reports/d4/8d4v ftp://data.pdbj.org/pub/pdb/validation_reports/d4/8d4v
ftp://data.pdbj.org/pub/pdb/validation_reports/d4/8d4v リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / Plasmid details: Purified from human sputum / 組織: Neutrophil / 参照: UniProt: P08311
Homo sapiens (ヒト) / Plasmid details: Purified from human sputum / 組織: Neutrophil / 参照: UniProt: P08311 Staphylococcus aureus subsp. aureus Mu50 (黄色ブドウ球菌)
Staphylococcus aureus subsp. aureus Mu50 (黄色ブドウ球菌)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-ID / 波長: 1 Å
/ ビームライン: 22-ID / 波長: 1 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj
















