| 登録情報 | データベース: PDB / ID: 8cyo
|
|---|
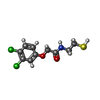
| タイトル | Nurr1 Covalently Bound to a Synthetic Ligand, 10.25, via a Disulfide Bond |
|---|
 要素 要素 | Nuclear receptor subfamily 4 group A member 2 |
|---|
 キーワード キーワード | TRANSCRIPTION / Disulfide Tethering |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
general adaptation syndrome / habenula development / cellular response to corticotropin-releasing hormone stimulus / central nervous system projection neuron axonogenesis / nuclear glucocorticoid receptor binding / regulation of dopamine metabolic process / regulation of respiratory gaseous exchange / midbrain dopaminergic neuron differentiation / dopaminergic neuron differentiation / neuron maturation ...general adaptation syndrome / habenula development / cellular response to corticotropin-releasing hormone stimulus / central nervous system projection neuron axonogenesis / nuclear glucocorticoid receptor binding / regulation of dopamine metabolic process / regulation of respiratory gaseous exchange / midbrain dopaminergic neuron differentiation / dopaminergic neuron differentiation / neuron maturation / central nervous system neuron differentiation / dopamine biosynthetic process / fat cell differentiation / negative regulation of apoptotic signaling pathway / canonical Wnt signaling pathway / nuclear retinoid X receptor binding / response to amphetamine / adult locomotory behavior / post-embryonic development / SUMOylation of intracellular receptors / beta-catenin binding / Nuclear Receptor transcription pathway / nuclear receptor activity / neuron migration / sequence-specific double-stranded DNA binding / cellular response to oxidative stress / neuron apoptotic process / DNA-binding transcription activator activity, RNA polymerase II-specific / transcription regulator complex / negative regulation of neuron apoptotic process / transcription by RNA polymerase II / DNA-binding transcription factor activity, RNA polymerase II-specific / response to hypoxia / nuclear speck / RNA polymerase II cis-regulatory region sequence-specific DNA binding / protein heterodimerization activity / DNA-templated transcription / regulation of transcription by RNA polymerase II / chromatin / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / protein-containing complex / DNA binding / zinc ion binding / nucleoplasm / nucleus / cytoplasm類似検索 - 分子機能 Orphan nuclear receptor, NURR type / Orphan nuclear receptor / Nuclear hormone receptor / Nuclear hormones receptors DNA-binding region signature. / Zinc finger, nuclear hormone receptor-type / Double treble clef zinc finger, C4 type / Nuclear hormone receptors DNA-binding domain profile. / c4 zinc finger in nuclear hormone receptors / Nuclear hormone receptor, ligand-binding domain / Nuclear hormone receptor-like domain superfamily ...Orphan nuclear receptor, NURR type / Orphan nuclear receptor / Nuclear hormone receptor / Nuclear hormones receptors DNA-binding region signature. / Zinc finger, nuclear hormone receptor-type / Double treble clef zinc finger, C4 type / Nuclear hormone receptors DNA-binding domain profile. / c4 zinc finger in nuclear hormone receptors / Nuclear hormone receptor, ligand-binding domain / Nuclear hormone receptor-like domain superfamily / Ligand-binding domain of nuclear hormone receptor / Nuclear receptor (NR) ligand-binding (LBD) domain profile. / Ligand binding domain of hormone receptors / Zinc finger, NHR/GATA-type類似検索 - ドメイン・相同性 Chem-OBJ / Nuclear receptor subfamily 4 group A member 2類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.41 Å 分子置換 / 解像度: 2.41 Å |
|---|
 データ登録者 データ登録者 | Bruning, J.M. / Liu, J. / England, P.M. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Human Genome Research Institute (NIH/NHGRI) | |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Nurr1 Covalently Bound to a Synthetic Ligand, 10.25, via a Disulfide Bond
著者: Holder, P. |
|---|
| 履歴 | | 登録 | 2022年5月24日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2024年1月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年11月13日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.41 Å
分子置換 / 解像度: 2.41 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8cyo.cif.gz
8cyo.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8cyo.ent.gz
pdb8cyo.ent.gz PDB形式
PDB形式 8cyo.json.gz
8cyo.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8cyo_validation.pdf.gz
8cyo_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8cyo_full_validation.pdf.gz
8cyo_full_validation.pdf.gz 8cyo_validation.xml.gz
8cyo_validation.xml.gz 8cyo_validation.cif.gz
8cyo_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/cy/8cyo
https://data.pdbj.org/pub/pdb/validation_reports/cy/8cyo ftp://data.pdbj.org/pub/pdb/validation_reports/cy/8cyo
ftp://data.pdbj.org/pub/pdb/validation_reports/cy/8cyo
 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: NR4A2, NOT, NURR1, TINUR / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: NR4A2, NOT, NURR1, TINUR / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 8.3.1 / 波長: 1.1158 Å
/ ビームライン: 8.3.1 / 波長: 1.1158 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj