| 登録情報 | データベース: PDB / ID: 8ccx
|
|---|
| タイトル | Human SOD1 in complex with S-XL6 cross-linker |
|---|
 要素 要素 | Superoxide dismutase [Cu-Zn] |
|---|
 キーワード キーワード | OXIDOREDUCTASE / SOD1 / Amyotrophic Lateral Sclerosis / ALS / pharmacological chaperone / cross-linking / stabilization |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
action potential initiation / response to antipsychotic drug / neurofilament cytoskeleton organization / response to carbon monoxide / protein phosphatase 2B binding / dense core granule / relaxation of vascular associated smooth muscle / anterograde axonal transport / regulation of organ growth / response to superoxide ...action potential initiation / response to antipsychotic drug / neurofilament cytoskeleton organization / response to carbon monoxide / protein phosphatase 2B binding / dense core granule / relaxation of vascular associated smooth muscle / anterograde axonal transport / regulation of organ growth / response to superoxide / regulation of T cell differentiation in thymus / positive regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / peripheral nervous system myelin maintenance / retina homeostasis / auditory receptor cell stereocilium organization / hydrogen peroxide biosynthetic process / cellular response to potassium ion / retrograde axonal transport / superoxide anion generation / regulation of GTPase activity / myeloid cell homeostasis / response to copper ion / superoxide metabolic process / muscle cell cellular homeostasis / superoxide dismutase / heart contraction / Detoxification of Reactive Oxygen Species / superoxide dismutase activity / cellular response to ATP / negative regulation of reproductive process / negative regulation of developmental process / cellular response to cadmium ion / transmission of nerve impulse / regulation of multicellular organism growth / ectopic germ cell programmed cell death / response to axon injury / neuronal action potential / ovarian follicle development / positive regulation of superoxide anion generation / axon cytoplasm / glutathione metabolic process / embryo implantation / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / dendrite cytoplasm / removal of superoxide radicals / reactive oxygen species metabolic process / positive regulation of phagocytosis / response to amphetamine / thymus development / positive regulation of cytokine production / placenta development / regulation of mitochondrial membrane potential / determination of adult lifespan / locomotory behavior / response to nutrient levels / response to hydrogen peroxide / sensory perception of sound / mitochondrial intermembrane space / small GTPase binding / regulation of blood pressure / negative regulation of inflammatory response / peroxisome / Platelet degranulation / protein-folding chaperone binding / response to heat / cytoplasmic vesicle / response to ethanol / spermatogenesis / gene expression / negative regulation of neuron apoptotic process / intracellular iron ion homeostasis / lysosome / positive regulation of MAPK cascade / positive regulation of apoptotic process / mitochondrial matrix / response to xenobiotic stimulus / copper ion binding / neuronal cell body / apoptotic process / protein homodimerization activity / protein-containing complex / mitochondrion / extracellular space / extracellular exosome / extracellular region / zinc ion binding / nucleoplasm / identical protein binding / nucleus / cytoplasm / cytosol類似検索 - 分子機能 Copper/Zinc superoxide dismutase signature 1. / Superoxide dismutase (Cu/Zn) / superoxide dismutase copper chaperone / Superoxide dismutase, copper/zinc, binding site / Copper/Zinc superoxide dismutase signature 2. / Superoxide dismutase, copper/zinc binding domain / Copper/zinc superoxide dismutase (SODC) / Superoxide dismutase-like, copper/zinc binding domain superfamily類似検索 - ドメイン・相同性 COPPER (II) ION / butane-1,4-dithiol / Superoxide dismutase [Cu-Zn]類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.665 Å 分子置換 / 解像度: 1.665 Å |
|---|
 データ登録者 データ登録者 | Antonyuk, S.V. / Hossain, A. / Agar, J.N. / Hasnain, S.S. |
|---|
| 資金援助 |  米国, 3件 米国, 3件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of Neurological Disorders and Stroke (NIH/NINDS) | R01NS065263 |  米国 米国 | | National Science Foundation (NSF, United States) | MCB-1517290 |  米国 米国 | | National Science Foundation (NSF, United States) | CHE-1905214 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Plos Biol. / 年: 2024 ジャーナル: Plos Biol. / 年: 2024
タイトル: Evaluating protein cross-linking as a therapeutic strategy to stabilize SOD1 variants in a mouse model of familial ALS.
著者: Hossain, M.A. / Sarin, R. / Donnelly, D.P. / Miller, B.C. / Weiss, A. / McAlary, L. / Antonyuk, S.V. / Salisbury, J.P. / Amin, J. / Conway, J.B. / Watson, S.S. / Winters, J.N. / Xu, Y. / ...著者: Hossain, M.A. / Sarin, R. / Donnelly, D.P. / Miller, B.C. / Weiss, A. / McAlary, L. / Antonyuk, S.V. / Salisbury, J.P. / Amin, J. / Conway, J.B. / Watson, S.S. / Winters, J.N. / Xu, Y. / Alam, N. / Brahme, R.R. / Shahbazian, H. / Sivasankar, D. / Padmakumar, S. / Sattarova, A. / Ponmudiyan, A.C. / Gawde, T. / Verrill, D.E. / Yang, W. / Kannapadi, S. / Plant, L.D. / Auclair, J.R. / Makowski, L. / Petsko, G.A. / Ringe, D. / Agar, N.Y.R. / Greenblatt, D.J. / Ondrechen, M.J. / Chen, Y. / Yerbury, J.J. / Manetsch, R. / Hasnain, S.S. / Brown Jr., R.H. / Agar, J.N. |
|---|
| 履歴 | | 登録 | 2023年1月27日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2023年12月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年2月7日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2024年10月23日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 1.665 Å
分子置換 / 解像度: 1.665 Å  データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Plos Biol. / 年: 2024
ジャーナル: Plos Biol. / 年: 2024 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8ccx.cif.gz
8ccx.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8ccx.ent.gz
pdb8ccx.ent.gz PDB形式
PDB形式 8ccx.json.gz
8ccx.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8ccx_validation.pdf.gz
8ccx_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8ccx_full_validation.pdf.gz
8ccx_full_validation.pdf.gz 8ccx_validation.xml.gz
8ccx_validation.xml.gz 8ccx_validation.cif.gz
8ccx_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/cc/8ccx
https://data.pdbj.org/pub/pdb/validation_reports/cc/8ccx ftp://data.pdbj.org/pub/pdb/validation_reports/cc/8ccx
ftp://data.pdbj.org/pub/pdb/validation_reports/cc/8ccx

 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: SOD1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: SOD1 / 発現宿主: 



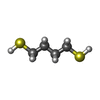







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E+ / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU FR-E+ / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj











