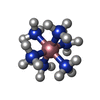
登録構造単位
A: DNA (5'-D(*(8OG)P*CP*AP*TP*GP*CP*T)-3')
B: DNA (5'-D(*(8OG)P*CP*AP*TP*GP*CP*T)-3')
ヘテロ分子 概要 構成要素の詳細
分子量 (理論値) 分子数 合計 (水以外) 4,903 6 ポリマ- 4,259 2 非ポリマー 644 4 水 847 47
1
A: DNA (5'-D(*(8OG)P*CP*AP*TP*GP*CP*T)-3')
ヘテロ分子
A: DNA (5'-D(*(8OG)P*CP*AP*TP*GP*CP*T)-3')
ヘテロ分子 概要 構成要素の詳細 対称操作
分子量 (理論値) 分子数 合計 (水以外) 5,548 10 ポリマ- 4,259 2 非ポリマー 1,289 8 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 3_554 -x,y,-z-1/2 1
2
B: DNA (5'-D(*(8OG)P*CP*AP*TP*GP*CP*T)-3')
B: DNA (5'-D(*(8OG)P*CP*AP*TP*GP*CP*T)-3') 概要 構成要素の詳細 対称操作
分子量 (理論値) 分子数 合計 (水以外) 4,259 2 ポリマ- 4,259 2 非ポリマー 0 0 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 3_554 -x,y,-z-1/2 1
単位格子 Length a, b, c (Å) 22.035, 58.846, 45.488 Angle α, β, γ (deg.) 90.000, 90.000, 90.000 Int Tables number 20 Space group name H-M C2221 Space group name Hall C2c2 Symmetry operation #1 : x,y,z#2 : x,-y,-z#3 : -x,y,-z+1/2#4 : -x,-y,z+1/2#5 : x+1/2,y+1/2,z#6 : x+1/2,-y+1/2,-z#7 : -x+1/2,y+1/2,-z+1/2#8 : -x+1/2,-y+1/2,z+1/2
Components on special symmetry positions ID モデル 要素 1 1 A -102-NCO
2 1 A -102-NCO
3 1 A -102-NCO
4 1 B -129-HOH
5 1 B -132-HOH
非結晶学的対称性 (NCS) NCSドメイン ID Ens-ID 詳細 d_1ens_1(chain "A" and resid 1 through 5)d_2ens_1(chain "B" and resid 1 through 5)
NCSドメイン領域 Dom-ID Component-ID Ens-ID Beg label comp-ID End label comp-ID Label asym-ID d_11 ens_18OGDGAd_21 ens_18OGDGB
NCS oper: (Code: givenMatrix: (0.998957069471, -0.0350448733463, -0.0292682456986), (-0.0356383850152, -0.99916442527, -0.0200089177104), (-0.0285425799052, 0.0210311228082, -0.999371308877) ... NCS oper Code Matrix (-0.0356383850152, -0.99916442527, -0.0200089177104), (-0.0285425799052, 0.0210311228082, -0.999371308877)ベクター -0.822621414885, -8.91881069764, -22.6150054217)
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 X線回折 /
X線回折 /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8baf.cif.gz
8baf.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8baf.ent.gz
pdb8baf.ent.gz PDB形式
PDB形式 8baf.json.gz
8baf.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8baf_validation.pdf.gz
8baf_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8baf_full_validation.pdf.gz
8baf_full_validation.pdf.gz 8baf_validation.xml.gz
8baf_validation.xml.gz 8baf_validation.cif.gz
8baf_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ba/8baf
https://data.pdbj.org/pub/pdb/validation_reports/ba/8baf ftp://data.pdbj.org/pub/pdb/validation_reports/ba/8baf
ftp://data.pdbj.org/pub/pdb/validation_reports/ba/8baf リンク
リンク 集合体
集合体


 要素
要素 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU PhotonJet-R / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU PhotonJet-R / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj