[English] 日本語
 Yorodumi
Yorodumi- PDB-7zyi: Structure of the human sodium/bile acid cotransporter (NTCP) in c... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7zyi | ||||||
|---|---|---|---|---|---|---|---|
| Title | Structure of the human sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody | ||||||
 Components Components |
| ||||||
 Keywords Keywords | TRANSPORT PROTEIN / NTCP / membrane protein / transport / sodium/bile acid cotransporter | ||||||
| Function / homology |  Function and homology information Function and homology informationbile acid:sodium symporter activity / bile acid transmembrane transporter activity / bile acid and bile salt transport / Recycling of bile acids and salts / response to nutrient levels / response to estrogen / cellular response to xenobiotic stimulus / virus receptor activity / basolateral plasma membrane / response to ethanol / plasma membrane Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human)  | ||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 2.88 Å | ||||||
 Authors Authors | Liu, H. / Irobalieva, R.N. / Bang-Sorensen, R. / Nosol, K. / Mukherjee, S. / Agrawal, P. / Stieger, B. / Kossiakoff, A.A. / Locher, K.P. | ||||||
| Funding support |  Switzerland, 1items Switzerland, 1items
| ||||||
 Citation Citation |  Journal: Cell Res / Year: 2022 Journal: Cell Res / Year: 2022Title: Structure of human NTCP reveals the basis of recognition and sodium-driven transport of bile salts into the liver. Authors: Hongtao Liu / Rossitza N Irobalieva / Rose Bang-Sørensen / Kamil Nosol / Somnath Mukherjee / Parth Agrawal / Bruno Stieger / Anthony A Kossiakoff / Kaspar P Locher /   | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7zyi.cif.gz 7zyi.cif.gz | 180.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7zyi.ent.gz pdb7zyi.ent.gz | 138.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7zyi.json.gz 7zyi.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/zy/7zyi https://data.pdbj.org/pub/pdb/validation_reports/zy/7zyi ftp://data.pdbj.org/pub/pdb/validation_reports/zy/7zyi ftp://data.pdbj.org/pub/pdb/validation_reports/zy/7zyi | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  15024MC M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
-Protein , 1 types, 1 molecules A
| #1: Protein | Mass: 38149.949 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: SLC10A1, NTCP, GIG29 / Cell (production host): HEK293 / Production host: Homo sapiens (human) / Gene: SLC10A1, NTCP, GIG29 / Cell (production host): HEK293 / Production host:  Homo sapiens (human) / References: UniProt: Q14973 Homo sapiens (human) / References: UniProt: Q14973 |
|---|
-Antibody , 3 types, 3 molecules HLK
| #2: Antibody | Mass: 25582.521 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
|---|---|
| #3: Antibody | Mass: 23417.971 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
| #4: Antibody | Mass: 13118.386 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
-Non-polymers , 4 types, 7 molecules 
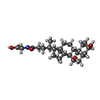





| #5: Chemical | | #6: Chemical | #7: Chemical | #8: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component | Name: sodium/bile acid cotransporter (NTCP) in complex with Fab and nanobody Type: COMPLEX / Entity ID: #1-#4 / Source: RECOMBINANT |
|---|---|
| Molecular weight | Value: 0.11 MDa / Experimental value: NO |
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Source (recombinant) | Organism:  Homo sapiens (human) / Cell: HEK293 Homo sapiens (human) / Cell: HEK293 |
| Buffer solution | pH: 7.4 |
| Specimen | Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES |
| Specimen support | Grid material: COPPER / Grid mesh size: 300 divisions/in. / Grid type: Quantifoil R1.2/1.3 |
| Vitrification | Instrument: FEI VITROBOT MARK IV / Cryogen name: ETHANE-PROPANE / Humidity: 100 % |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: TFS KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal defocus max: 2400 nm / Nominal defocus min: 600 nm |
| Specimen holder | Cryogen: NITROGEN |
| Image recording | Average exposure time: 2 sec. / Electron dose: 64 e/Å2 / Film or detector model: GATAN K3 BIOQUANTUM (6k x 4k) / Num. of grids imaged: 1 / Num. of real images: 13208 |
- Processing
Processing
| Software | Name: PHENIX / Version: 1.20.1_4487: / Classification: refinement | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EM software |
| ||||||||||||||||||||||||
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3D reconstruction | Resolution: 2.88 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 161093 / Symmetry type: POINT | ||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller


 PDBj
PDBj













