+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7z9l | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Phen-DC3 intercalation causes hybrid-to-antiparallel transformation of human telomeric DNA G-quadruplex | ||||||||||||||||||||||||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / G Quadruplex / ligand intercalation / NMR spectroscopy / Mass spectrometry | 機能・相同性 | Chem-PQ3 / DNA / DNA (> 10) |  機能・相同性情報 機能・相同性情報生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)手法 | 溶液NMR / simulated annealing |  データ登録者 データ登録者Ghosh, A. / Trajkovski, M. / Teulade-Fichou, M.P. / Gabelica, V. / Plavec, J. | 資金援助 | European Union, |  スロベニア, スロベニア,  フランス, 4件 フランス, 4件
 引用 引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2022 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2022タイトル: Phen-DC 3 Induces Refolding of Human Telomeric DNA into a Chair-Type Antiparallel G-Quadruplex through Ligand Intercalation. 著者: Ghosh, A. / Trajkovski, M. / Teulade-Fichou, M.P. / Gabelica, V. / Plavec, J. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7z9l.cif.gz 7z9l.cif.gz | 166.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7z9l.ent.gz pdb7z9l.ent.gz | 133.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7z9l.json.gz 7z9l.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7z9l_validation.pdf.gz 7z9l_validation.pdf.gz | 449.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7z9l_full_validation.pdf.gz 7z9l_full_validation.pdf.gz | 494.9 KB | 表示 | |
| XML形式データ |  7z9l_validation.xml.gz 7z9l_validation.xml.gz | 8.5 KB | 表示 | |
| CIF形式データ |  7z9l_validation.cif.gz 7z9l_validation.cif.gz | 12.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/z9/7z9l https://data.pdbj.org/pub/pdb/validation_reports/z9/7z9l ftp://data.pdbj.org/pub/pdb/validation_reports/z9/7z9l ftp://data.pdbj.org/pub/pdb/validation_reports/z9/7z9l | HTTPS FTP |
-関連構造データ
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 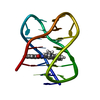
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
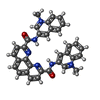
| #1: DNA鎖 | 分子量: 7287.690 Da / 分子数: 1 / 由来タイプ: 合成 詳細: The DNA is bound to a small molecule named N2,N9-bis(1-methylquinolin-3-yl)-1,10-phenanthroline-2,9-dicarboxamide(Phen-DC3). Three letter code (PQ3). 由来: (合成)  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|---|
| #2: 化合物 | ChemComp-PQ3 / |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||
| 試料状態 | 詳細: 20 mM KCl, 70 mM potassium phosphate buffer with pH 7.0 containing 95% H2O+ 5% D2O and 100% D2O イオン強度: 70 mM Label: 20 mM KCl, 70 mM potassium phosphate buffer with pH 7.0 containing 5% D2O pH: 7 / 圧: 1 atm / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: 497 distance, 24 Planarity, 23 dihedral angle, 16 H bonding | ||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj







































 HSQC
HSQC