+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7z9c | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E.coli gyrase holocomplex with 217 bp DNA and albicidin | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | ISOMERASE / type II topoisomerase / antibiotic / albicidin / DNA gyrase | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of DNA-templated DNA replication / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA negative supercoiling activity / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / ATP-dependent activity, acting on DNA / DNA-templated DNA replication / chromosome / response to xenobiotic stimulus ...negative regulation of DNA-templated DNA replication / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA negative supercoiling activity / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / ATP-dependent activity, acting on DNA / DNA-templated DNA replication / chromosome / response to xenobiotic stimulus / response to antibiotic / DNA-templated transcription / DNA binding / ATP binding / metal ion binding / identical protein binding / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 生物種 |   Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.06 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Ghilarov, D. / Heddle, J.G.H. / Suessmuth, R. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 資金援助 |  英国, 英国,  ポーランド, 2件 ポーランド, 2件
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Catal / 年: 2023 ジャーナル: Nat Catal / 年: 2023タイトル: Molecular mechanism of topoisomerase poisoning by the peptide antibiotic albicidin. 著者: Elizabeth Michalczyk / Kay Hommernick / Iraj Behroz / Marcel Kulike / Zuzanna Pakosz-Stępień / Lukasz Mazurek / Maria Seidel / Maria Kunert / Karine Santos / Holger von Moeller / Bernhard ...著者: Elizabeth Michalczyk / Kay Hommernick / Iraj Behroz / Marcel Kulike / Zuzanna Pakosz-Stępień / Lukasz Mazurek / Maria Seidel / Maria Kunert / Karine Santos / Holger von Moeller / Bernhard Loll / John B Weston / Andi Mainz / Jonathan G Heddle / Roderich D Süssmuth / Dmitry Ghilarov /    要旨: The peptide antibiotic albicidin is a DNA topoisomerase inhibitor with low-nanomolar bactericidal activity towards fluoroquinolone-resistant Gram-negative pathogens. However, its mode of action is ...The peptide antibiotic albicidin is a DNA topoisomerase inhibitor with low-nanomolar bactericidal activity towards fluoroquinolone-resistant Gram-negative pathogens. However, its mode of action is poorly understood. We determined a 2.6 Å resolution cryoelectron microscopy structure of a ternary complex between topoisomerase DNA gyrase, a 217 bp double-stranded DNA fragment and albicidin. Albicidin employs a dual binding mechanism where one end of the molecule obstructs the crucial gyrase dimer interface, while the other intercalates between the fragments of cleaved DNA substrate. Thus, albicidin efficiently locks DNA gyrase, preventing it from religating DNA and completing its catalytic cycle. Two additional structures of this trapped state were determined using synthetic albicidin analogues that demonstrate improved solubility, and activity against a range of gyrase variants and topoisomerase IV. The extraordinary promiscuity of the DNA-intercalating region of albicidins and their excellent performance against fluoroquinolone-resistant bacteria holds great promise for the development of last-resort antibiotics. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7z9c.cif.gz 7z9c.cif.gz | 686.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7z9c.ent.gz pdb7z9c.ent.gz | 545.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7z9c.json.gz 7z9c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7z9c_validation.pdf.gz 7z9c_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7z9c_full_validation.pdf.gz 7z9c_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  7z9c_validation.xml.gz 7z9c_validation.xml.gz | 64.3 KB | 表示 | |
| CIF形式データ |  7z9c_validation.cif.gz 7z9c_validation.cif.gz | 98 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/z9/7z9c https://data.pdbj.org/pub/pdb/validation_reports/z9/7z9c ftp://data.pdbj.org/pub/pdb/validation_reports/z9/7z9c ftp://data.pdbj.org/pub/pdb/validation_reports/z9/7z9c | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-DNA gyrase subunit ... , 2種, 4分子 ACBD
| #1: タンパク質 | 分子量: 97854.305 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  株: K12 / 遺伝子: gyrA, hisW, nalA, parD, b2231, JW2225 / 発現宿主:  参照: UniProt: P0AES4, DNA topoisomerase (ATP-hydrolysing) #2: タンパク質 | 分子量: 90891.734 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  株: K12 遺伝子: gyrB, acrB, cou, himB, hisU, nalC, parA, pcbA, b3699, JW5625 発現宿主:  参照: UniProt: P0AES6, DNA topoisomerase (ATP-hydrolysing) |
|---|
-DNA鎖 , 4種, 4分子 EFGH
| #3: DNA鎖 | 分子量: 4335.827 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Escherichia phage Mu (ファージ) / 発現宿主: Escherichia phage Mu (ファージ) / 発現宿主:  |
|---|---|
| #4: DNA鎖 | 分子量: 4321.856 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Escherichia phage Mu (ファージ) / 発現宿主: Escherichia phage Mu (ファージ) / 発現宿主:  |
| #5: DNA鎖 | 分子量: 5444.558 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Escherichia phage Mu (ファージ) / 発現宿主: Escherichia phage Mu (ファージ) / 発現宿主:  |
| #6: DNA鎖 | 分子量: 5487.561 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Escherichia phage Mu (ファージ) / 発現宿主: Escherichia phage Mu (ファージ) / 発現宿主:  |
-非ポリマー , 3種, 13分子 
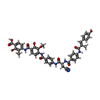



| #7: 化合物 | | #8: 化合物 | ChemComp-BWH / | #9: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | ||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||
| 緩衝液 | pH: 8 | ||||||||||||||||||||||||
| 緩衝液成分 | 濃度: 20 mM / 名称: HEPES | ||||||||||||||||||||||||
| 試料 | 濃度: 12 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 95 % / 凍結前の試料温度: 283 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 900 nm |
| 撮影 | 電子線照射量: 40 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.19.2_4158: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア | 名称: PHENIX / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2040353 | ||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.06 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 23435 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj








































