+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7vzg | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Acidobacteria homodimeric reaction center bound with cytochrome c (the larger form) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PHOTOSYNTHESIS / membrane protein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thylakoid / iron-sulfur cluster binding / photosynthesis / electron transfer activity / iron ion binding / heme binding / metal ion binding / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.61 Å | ||||||
 データ登録者 データ登録者 | Huang, G.Q. / Dong, S.S. / Qin, X.C. / Sui, S.F. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of the Acidobacteria homodimeric reaction center bound with cytochrome c. 著者: Shishang Dong / Guoqiang Huang / Changhui Wang / Jiajia Wang / Sen-Fang Sui / Xiaochun Qin /  要旨: Photosynthesis converts light energy to chemical energy to fuel life on earth. Light energy is harvested by antenna pigments and transferred to reaction centers (RCs) to drive the electron transfer ...Photosynthesis converts light energy to chemical energy to fuel life on earth. Light energy is harvested by antenna pigments and transferred to reaction centers (RCs) to drive the electron transfer (ET) reactions. Here, we present cryo-electron microscopy (cryo-EM) structures of two forms of the RC from the microaerophilic Chloracidobacterium thermophilum (CabRC): one containing 10 subunits, including two different cytochromes; and the other possessing two additional subunits, PscB and PscZ. The larger form contained 2 Zn-bacteriochlorophylls, 16 bacteriochlorophylls, 10 chlorophylls, 2 lycopenes, 2 hemes, 3 FeS clusters, 12 lipids, 2 Ca ions and 6 water molecules, revealing a type I RC with an ET chain involving two hemes and a hybrid antenna containing bacteriochlorophylls and chlorophylls. Our results provide a structural basis for understanding the excitation energy and ET within the CabRC and offer evolutionary insights into the origin and adaptation of photosynthetic RCs. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7vzg.cif.gz 7vzg.cif.gz | 497.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7vzg.ent.gz pdb7vzg.ent.gz | 401.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7vzg.json.gz 7vzg.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7vzg_validation.pdf.gz 7vzg_validation.pdf.gz | 2.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7vzg_full_validation.pdf.gz 7vzg_full_validation.pdf.gz | 3.2 MB | 表示 | |
| XML形式データ |  7vzg_validation.xml.gz 7vzg_validation.xml.gz | 111.9 KB | 表示 | |
| CIF形式データ |  7vzg_validation.cif.gz 7vzg_validation.cif.gz | 149.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/vz/7vzg https://data.pdbj.org/pub/pdb/validation_reports/vz/7vzg ftp://data.pdbj.org/pub/pdb/validation_reports/vz/7vzg ftp://data.pdbj.org/pub/pdb/validation_reports/vz/7vzg | HTTPS FTP |
-関連構造データ
| 関連構造データ |  32228MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 5種, 6分子 AaCcBD
| #1: タンパク質 | 分子量: 98537.555 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: G2LDR8 #2: タンパク質 | | 分子量: 22369.820 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: G2LDR4 #7: タンパク質 | | 分子量: 15664.669 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: G2LDR3 #8: タンパク質 | | 分子量: 8199.330 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: A8DJF8 #9: タンパク質 | | 分子量: 8622.735 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: G2LHG2 |
|---|
-タンパク質・ペプチド , 4種, 8分子 EeFfGgHh
| #3: タンパク質・ペプチド | 分子量: 3684.458 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: G2LK98 #4: タンパク質・ペプチド | 分子量: 3885.684 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: G2LEN5 #5: タンパク質・ペプチド | 分子量: 4293.273 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア)参照: UniProt: G2LJ20 #6: タンパク質・ペプチド | 分子量: 1635.006 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア) |
|---|
-非ポリマー , 12種, 87分子 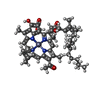



















| #10: 化合物 | | #11: 化合物 | ChemComp-BCL / #12: 化合物 | ChemComp-CLA / #13: 化合物 | #14: 化合物 | #15: 化合物 | ChemComp-85I / [( #16: 化合物 | ChemComp-UNL / 分子量: 663.906 Da / 分子数: 32 / 由来タイプ: 合成 / タイプ: SUBJECT OF INVESTIGATION #17: 化合物 | ChemComp-85N / [( #18: 化合物 | #19: 化合物 | #20: 化合物 | #21: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: CabRC complex, the larger form / タイプ: COMPLEX / Entity ID: #1-#9 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  Chloracidobacterium thermophilum (バクテリア) Chloracidobacterium thermophilum (バクテリア) |
| 緩衝液 | pH: 7.6 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 500 nm / Cs: 2.7 mm |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION |
|---|---|
| 3次元再構成 | 解像度: 2.61 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 52612 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj




























