+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7sut | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Light harvesting phycobiliprotein HaPE645 from the cryptophyte Hemiselmis andersenii CCMP644 | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | PHOTOSYNTHESIS / phycobiliprotein / antenna / light harvesting / cryptophyte / algae / globin / CALM / CaRSP / phycoerythrin / Hemiselmis | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Hemiselmis andersenii (真核生物) Hemiselmis andersenii (真核生物) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.49 Å 分子置換 / 解像度: 1.49 Å | ||||||||||||
 データ登録者 データ登録者 | Rathbone, H.W. / Michie, K.A. / Laos, A.L. / Curmi, P.M.G. | ||||||||||||
| 資金援助 |  オーストラリア, 3件 オーストラリア, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: Molecular dissection of the soluble photosynthetic antenna from the cryptophyte alga Hemiselmis andersenii. 著者: Rathbone, H.W. / Laos, A.J. / Michie, K.A. / Iranmanesh, H. / Biazik, J. / Goodchild, S.C. / Thordarson, P. / Green, B.R. / Curmi, P.M.G. #1:  ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2012 ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2012タイトル: Towards automated crystallographic structure refinement with phenix.refine. 著者: Afonine, P.V. / Grosse-Kunstleve, R.W. / Echols, N. / Headd, J.J. / Moriarty, N.W. / Mustyakimov, M. / Terwilliger, T.C. / Urzhumtsev, A. / Zwart, P.H. / Adams, P.D. #2:  ジャーナル: J Synchrotron Radiat / 年: 2002 ジャーナル: J Synchrotron Radiat / 年: 2002タイトル: Blu-Ice and the Distributed Control System: software for data acquisition and instrument control at macromolecular crystallography beamlines. 著者: McPhillips, T.M. / McPhillips, S.E. / Chiu, H.J. / Cohen, A.E. / Deacon, A.M. / Ellis, P.J. / Garman, E. / Gonzalez, A. / Sauter, N.K. / Phizackerley, R.P. / Soltis, S.M. / Kuhn, P. #3:  ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2004 ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2004タイトル: Coot: model-building tools for molecular graphics. 著者: Emsley, P. / Cowtan, K. #4:  ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2011 ジャーナル: Acta Crystallogr D Biol Crystallogr / 年: 2011タイトル: Overview of the CCP4 suite and current developments. 著者: Winn, M.D. / Ballard, C.C. / Cowtan, K.D. / Dodson, E.J. / Emsley, P. / Evans, P.R. / Keegan, R.M. / Krissinel, E.B. / Leslie, A.G. / McCoy, A. / McNicholas, S.J. / Murshudov, G.N. / Pannu, N. ...著者: Winn, M.D. / Ballard, C.C. / Cowtan, K.D. / Dodson, E.J. / Emsley, P. / Evans, P.R. / Keegan, R.M. / Krissinel, E.B. / Leslie, A.G. / McCoy, A. / McNicholas, S.J. / Murshudov, G.N. / Pannu, N.S. / Potterton, E.A. / Powell, H.R. / Read, R.J. / Vagin, A. / Wilson, K.S. #5:  ジャーナル: J Appl Crystallogr / 年: 2007 ジャーナル: J Appl Crystallogr / 年: 2007タイトル: Phaser crystallographic software. 著者: McCoy, A.J. / Grosse-Kunstleve, R.W. / Adams, P.D. / Winn, M.D. / Storoni, L.C. / Read, R.J. #6:  ジャーナル: J Synchrotron Radiat / 年: 2018 ジャーナル: J Synchrotron Radiat / 年: 2018タイトル: MX2: a high-flux undulator microfocus beamline serving both the chemical and macromolecular crystallography communities at the Australian Synchrotron. 著者: Aragao, D. / Aishima, J. / Cherukuvada, H. / Clarken, R. / Clift, M. / Cowieson, N.P. / Ericsson, D.J. / Gee, C.L. / Macedo, S. / Mudie, N. / Panjikar, S. / Price, J.R. / Riboldi-Tunnicliffe, ...著者: Aragao, D. / Aishima, J. / Cherukuvada, H. / Clarken, R. / Clift, M. / Cowieson, N.P. / Ericsson, D.J. / Gee, C.L. / Macedo, S. / Mudie, N. / Panjikar, S. / Price, J.R. / Riboldi-Tunnicliffe, A. / Rostan, R. / Williamson, R. / Caradoc-Davies, T.T. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7sut.cif.gz 7sut.cif.gz | 612.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7sut.ent.gz pdb7sut.ent.gz | 502.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7sut.json.gz 7sut.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7sut_validation.pdf.gz 7sut_validation.pdf.gz | 5.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7sut_full_validation.pdf.gz 7sut_full_validation.pdf.gz | 5.5 MB | 表示 | |
| XML形式データ |  7sut_validation.xml.gz 7sut_validation.xml.gz | 50.4 KB | 表示 | |
| CIF形式データ |  7sut_validation.cif.gz 7sut_validation.cif.gz | 64.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/su/7sut https://data.pdbj.org/pub/pdb/validation_reports/su/7sut ftp://data.pdbj.org/pub/pdb/validation_reports/su/7sut ftp://data.pdbj.org/pub/pdb/validation_reports/su/7sut | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 単位格子 |
|
- 要素
要素
-HaPE645 alpha- ... , 2種, 4分子 AECG
| #1: タンパク質 | 分子量: 8974.020 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Hemiselmis andersenii (真核生物) / 参照: UniProt: A0A7S0U215 Hemiselmis andersenii (真核生物) / 参照: UniProt: A0A7S0U215#3: タンパク質 | 分子量: 7795.986 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Hemiselmis andersenii (真核生物) / 株: CCMP644 Hemiselmis andersenii (真核生物) / 株: CCMP644 |
|---|
-タンパク質 , 1種, 4分子 BDFH
| #2: タンパク質 | 分子量: 18406.949 Da / 分子数: 4 / 由来タイプ: 天然 / 由来: (天然)  Hemiselmis andersenii (真核生物) / 参照: UniProt: U5T8W0 Hemiselmis andersenii (真核生物) / 参照: UniProt: U5T8W0 |
|---|
-非ポリマー , 8種, 476分子 


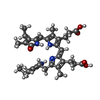











| #4: 化合物 | ChemComp-X2I / ( #5: 化合物 | #6: 化合物 | ChemComp-CL / | #7: 化合物 | ChemComp-AX9 / #8: 化合物 | ChemComp-PEB / #9: 化合物 | ChemComp-BTB / | #10: 化合物 | #11: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.36 Å3/Da / 溶媒含有率: 47.95 % |
|---|---|
| 結晶化 | 温度: 300 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 5.5 / 詳細: 0.1 M Bis-Tris + 25 % (w/v) PEG 3350 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Australian Synchrotron Australian Synchrotron  / ビームライン: MX2 / 波長: 0.9537 Å / ビームライン: MX2 / 波長: 0.9537 Å |
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2020年2月28日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9537 Å / 相対比: 1 |
| 反射 | 解像度: 1.49→115.75 Å / Num. obs: 151716 / % possible obs: 93.4 % / 冗長度: 3.6 % / Biso Wilson estimate: 19.89 Å2 / CC1/2: 0.998 / Rmerge(I) obs: 0.056 / Net I/σ(I): 9.9 |
| 反射 シェル | 解像度: 1.49→1.52 Å / 冗長度: 2.2 % / Rmerge(I) obs: 1.117 / Mean I/σ(I) obs: 0.9 / Num. unique obs: 4645 / CC1/2: 0.463 / % possible all: 58.3 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 4LMX 解像度: 1.49→41.43 Å / SU ML: 0.1629 / 交差検証法: FREE R-VALUE / σ(F): 1.37 / 位相誤差: 18.3653 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 26.94 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.49→41.43 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj








