登録情報 データベース : PDB / ID : 7pn9タイトル Evolved unspecific peroxygenase with A77L mutation in complex with lauric acid Aromatic peroxygenase キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / 生物種 Agrocybe aegerita (菌類)手法 / / / 解像度 : 1.85 Å データ登録者 Fernandez-Garcia, A. / Sanz-Aparicio, J. 資金援助 組織 認可番号 国 Spanish Ministry of Science, Innovation, and Universities Comunidad de Madrid Synergy CAM project Y2018/BIO-4738-EVOCHIMERA-CM
ジャーナル : Angew.Chem.Int.Ed.Engl. / 年 : 2023タイトル : Engineering a Highly Regioselective Fungal Peroxygenase for the Synthesis of Hydroxy Fatty Acids.著者: Gomez de Santos, P. / Gonzalez-Benjumea, A. / Fernandez-Garcia, A. / Aranda, C. / Wu, Y. / But, A. / Molina-Espeja, P. / Mate, D.M. / Gonzalez-Perez, D. / Zhang, W. / Kiebist, J. / Scheibner, ... 著者 : Gomez de Santos, P. / Gonzalez-Benjumea, A. / Fernandez-Garcia, A. / Aranda, C. / Wu, Y. / But, A. / Molina-Espeja, P. / Mate, D.M. / Gonzalez-Perez, D. / Zhang, W. / Kiebist, J. / Scheibner, K. / Hofrichter, M. / Swiderek, K. / Moliner, V. / Sanz-Aparicio, J. / Hollmann, F. / Gutierrez, A. / Alcalde, M. 履歴 登録 2021年9月6日 登録サイト / 処理サイト 改定 1.0 2023年1月25日 Provider / タイプ 改定 1.1 2023年2月1日 Group / カテゴリ / citation_authorItem _citation.page_first / _citation.page_last ... _citation.page_first / _citation.page_last / _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.identifier_ORCID / _citation_author.name 改定 1.2 2023年3月1日 Group / カテゴリ / Item / _citation.year改定 1.3 2024年2月7日 Group / Refinement descriptionカテゴリ / chem_comp_bond / pdbx_initial_refinement_model改定 1.4 2024年11月13日 Group カテゴリ / pdbx_modification_featureItem
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Agrocybe aegerita (菌類)
Agrocybe aegerita (菌類) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 1.85 Å
フーリエ合成 / 解像度: 1.85 Å  データ登録者
データ登録者 スペイン, 1件
スペイン, 1件  引用
引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2023
ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2023 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7pn9.cif.gz
7pn9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7pn9.ent.gz
pdb7pn9.ent.gz PDB形式
PDB形式 7pn9.json.gz
7pn9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7pn9_validation.pdf.gz
7pn9_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7pn9_full_validation.pdf.gz
7pn9_full_validation.pdf.gz 7pn9_validation.xml.gz
7pn9_validation.xml.gz 7pn9_validation.cif.gz
7pn9_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/pn/7pn9
https://data.pdbj.org/pub/pdb/validation_reports/pn/7pn9 ftp://data.pdbj.org/pub/pdb/validation_reports/pn/7pn9
ftp://data.pdbj.org/pub/pdb/validation_reports/pn/7pn9 リンク
リンク 集合体
集合体
 要素
要素

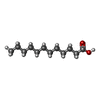
 Agrocybe aegerita (菌類) / 遺伝子: APO1 / 発現宿主:
Agrocybe aegerita (菌類) / 遺伝子: APO1 / 発現宿主:  Komagataella phaffii CBS 7435 (菌類) / 参照: UniProt: B9W4V6, unspecific peroxygenase
Komagataella phaffii CBS 7435 (菌類) / 参照: UniProt: B9W4V6, unspecific peroxygenase











 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALBA
ALBA  / ビームライン: XALOC / 波長: 0.97926 Å
/ ビームライン: XALOC / 波長: 0.97926 Å 解析
解析 フーリエ合成
フーリエ合成 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj







