+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7lna | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Infectious mammalian prion fibril (263K scrapie) | |||||||||
 要素 要素 | Major prion protein | |||||||||
 キーワード キーワード | PROTEIN FIBRIL / infectious mammalian prion / templating / glycosylated glycophophatidlyinositol-anchored amyloid / PIRIBS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of glutamate receptor signaling pathway / regulation of calcium ion import across plasma membrane / aspartic-type endopeptidase inhibitor activity / glycosaminoglycan binding / regulation of potassium ion transmembrane transport / negative regulation of interleukin-17 production / negative regulation of dendritic spine maintenance / type 5 metabotropic glutamate receptor binding / cupric ion binding / negative regulation of calcineurin-NFAT signaling cascade ...regulation of glutamate receptor signaling pathway / regulation of calcium ion import across plasma membrane / aspartic-type endopeptidase inhibitor activity / glycosaminoglycan binding / regulation of potassium ion transmembrane transport / negative regulation of interleukin-17 production / negative regulation of dendritic spine maintenance / type 5 metabotropic glutamate receptor binding / cupric ion binding / negative regulation of calcineurin-NFAT signaling cascade / negative regulation of interleukin-2 production / negative regulation of T cell receptor signaling pathway / cuprous ion binding / negative regulation of amyloid-beta formation / negative regulation of activated T cell proliferation / : / negative regulation of type II interferon production / positive regulation of protein targeting to membrane / side of membrane / inclusion body / cellular response to copper ion / neuron projection maintenance / negative regulation of protein phosphorylation / molecular condensate scaffold activity / molecular function activator activity / positive regulation of protein localization to plasma membrane / protein destabilization / protein homooligomerization / terminal bouton / cellular response to amyloid-beta / positive regulation of peptidyl-tyrosine phosphorylation / positive regulation of neuron apoptotic process / cellular response to xenobiotic stimulus / signaling receptor activity / amyloid-beta binding / microtubule binding / nuclear membrane / protease binding / response to oxidative stress / amyloid fibril formation / learning or memory / regulation of cell cycle / membrane raft / cell cycle / copper ion binding / dendrite / protein-containing complex binding / negative regulation of apoptotic process / Golgi apparatus / cell surface / endoplasmic reticulum / identical protein binding / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mesocricetus auratus (ネズミ) Mesocricetus auratus (ネズミ) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.14 Å | |||||||||
 データ登録者 データ登録者 | Kraus, A. / Hoyt, F. / Schwartz, C.L. / Hansen, B. / Hughson, A.G. / Artikis, E. / Race, B. / Caughey, B. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: High-resolution structure and strain comparison of infectious mammalian prions. 著者: Allison Kraus / Forrest Hoyt / Cindi L Schwartz / Bryan Hansen / Efrosini Artikis / Andrew G Hughson / Gregory J Raymond / Brent Race / Gerald S Baron / Byron Caughey /  要旨: Within the extensive range of self-propagating pathologic protein aggregates of mammals, prions are the most clearly infectious (e.g., ∼10 lethal doses per milligram). The structures of such lethal ...Within the extensive range of self-propagating pathologic protein aggregates of mammals, prions are the most clearly infectious (e.g., ∼10 lethal doses per milligram). The structures of such lethal assemblies of PrP molecules have been poorly understood. Here we report a near-atomic core structure of a brain-derived, fully infectious prion (263K strain). Cryo-electron microscopy showed amyloid fibrils assembled with parallel in-register intermolecular β sheets. Each monomer provides one rung of the ordered fibril core, with N-linked glycans and glycolipid anchors projecting outward. Thus, single monomers form the templating surface for incoming monomers at fibril ends, where prion growth occurs. Comparison to another prion strain (aRML) revealed major differences in fibril morphology but, like 263K, an asymmetric fibril cross-section without paired protofilaments. These findings provide structural insights into prion propagation, strains, species barriers, and membrane pathogenesis. This structure also helps frame considerations of factors influencing the relative transmissibility of other pathologic amyloids. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7lna.cif.gz 7lna.cif.gz | 76.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7lna.ent.gz pdb7lna.ent.gz | 57.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7lna.json.gz 7lna.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7lna_validation.pdf.gz 7lna_validation.pdf.gz | 976.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7lna_full_validation.pdf.gz 7lna_full_validation.pdf.gz | 977.3 KB | 表示 | |
| XML形式データ |  7lna_validation.xml.gz 7lna_validation.xml.gz | 29.9 KB | 表示 | |
| CIF形式データ |  7lna_validation.cif.gz 7lna_validation.cif.gz | 41.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ln/7lna https://data.pdbj.org/pub/pdb/validation_reports/ln/7lna ftp://data.pdbj.org/pub/pdb/validation_reports/ln/7lna ftp://data.pdbj.org/pub/pdb/validation_reports/ln/7lna | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 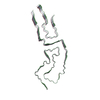
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCSアンサンブル:
|
- 要素
要素
| #1: タンパク質 | 分子量: 16264.101 Da / 分子数: 3 / 由来タイプ: 天然 詳細: Protease-resistant glycosylated, glycophophatidlyinositol-anchored infectious prion amyloid core (inclusive of residues 90-231). 由来: (天然)  Mesocricetus auratus (ネズミ) / 器官: brain / 参照: UniProt: P04273 Mesocricetus auratus (ネズミ) / 器官: brain / 参照: UniProt: P04273 |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: infectious mammalian prion fibril (263K scrapie) / タイプ: COMPLEX 詳細: Purified from brains of hamsters with clinical scrapie prion disease. Entity ID: all / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  Mesocricetus auratus (ネズミ) / 器官: brain / 組織: brain Mesocricetus auratus (ネズミ) / 器官: brain / 組織: brain |
| 緩衝液 | pH: 7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: Solarus 950 (Gatan, Pleasanton CA) / グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: C-flat-1.2/1.3 |
| 急速凍結 | 装置: LEICA EM GP / 凍結剤: ETHANE / 湿度: 90 % / 凍結前の試料温度: 295 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD |
| 試料ホルダ | 凍結剤: NITROGEN |
| 撮影 | 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0258 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 画像処理 | 詳細: Processing and reconstruction was conducted in Relion as per Scheres, Acta Cryst. (2020). D76, 94-101 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 337368 詳細: Filament start and end positions were picked manually in RELION. Particles were extracted with an interbox distance of 14.7A along the filament axis. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.14 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 15884 / アルゴリズム: FOURIER SPACE 詳細: Auto-refinement was then performed while optimizing the helical twist and rise. Auto-refinement with refinement of twist and rise yielded a final map with a twist of -0.847 degree and rise of ...詳細: Auto-refinement was then performed while optimizing the helical twist and rise. Auto-refinement with refinement of twist and rise yielded a final map with a twist of -0.847 degree and rise of 4.874 angstrom. Iterative cycles of CTF refinement, Bayesian polishing, and auto refinement were used until resolution estimates stabilized. Post processing in RELION was performed with a soft-edged mask representing 10% of the central Z length of the fibril. Sharpening was applied with a B-factor of -31 square angstrom. 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.1→114.45 Å / Cor.coef. Fo:Fc: 0.808 / ESU R: 0.544 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 39.924 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 3171 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj
