| 登録情報 | データベース: PDB / ID: 7a0i
|
|---|
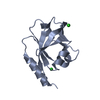
| タイトル | NMR structure of flagelliform spidroin (FlagSp) N-terminal domain from Trichonephila clavipes at pH 7.2 |
|---|
 要素 要素 | Flagelliform spidroin variant 1 |
|---|
 キーワード キーワード | STRUCTURAL PROTEIN / FLAGELLIFORM SPIDROIN / NT DOMAIN / pH 7.2 |
|---|
| 機能・相同性 | Spidroin, N-terminal / Major ampullate spidroin 1, spider silk protein 1, N-term / Spidroin, N-terminal domain superfamily / Flagelliform spidroin variant 1 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Nephila clavipes (クモ) Nephila clavipes (クモ) |
|---|
| 手法 | 溶液NMR / torsion angle dynamics / simulated annealing |
|---|
 データ登録者 データ登録者 | Sarr, M. / Kitoka, K. / Walsh-White, K.-A. / Kaldmae, M. / Landreh, M. / Rising, A. / Johansson, J. / Jaudzems, K. / Kronqvist, N. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2022 ジャーナル: J.Biol.Chem. / 年: 2022
タイトル: The dimerization mechanism of the N-terminal domain of spider silk proteins is conserved despite extensive sequence divergence.
著者: Sarr, M. / Kitoka, K. / Walsh-White, K.A. / Kaldmae, M. / Metlans, R. / Tars, K. / Mantese, A. / Shah, D. / Landreh, M. / Rising, A. / Johansson, J. / Jaudzems, K. / Kronqvist, N. |
|---|
| 履歴 | | 登録 | 2020年8月9日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2021年8月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年8月31日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Other / カテゴリ: pdbx_database_status / Item: _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.3 | 2024年6月19日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Nephila clavipes (クモ)
Nephila clavipes (クモ) データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2022
ジャーナル: J.Biol.Chem. / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7a0i.cif.gz
7a0i.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7a0i.ent.gz
pdb7a0i.ent.gz PDB形式
PDB形式 7a0i.json.gz
7a0i.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7a0i_validation.pdf.gz
7a0i_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7a0i_full_validation.pdf.gz
7a0i_full_validation.pdf.gz 7a0i_validation.xml.gz
7a0i_validation.xml.gz 7a0i_validation.cif.gz
7a0i_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/a0/7a0i
https://data.pdbj.org/pub/pdb/validation_reports/a0/7a0i ftp://data.pdbj.org/pub/pdb/validation_reports/a0/7a0i
ftp://data.pdbj.org/pub/pdb/validation_reports/a0/7a0i

 リンク
リンク 集合体
集合体
 要素
要素 Nephila clavipes (クモ) / 遺伝子: Flag / 発現宿主:
Nephila clavipes (クモ) / 遺伝子: Flag / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj gel filtration
gel filtration