[English] 日本語
 Yorodumi
Yorodumi- PDB-6yku: Neisseria gonorrhoeae Leucyl-tRNA Synthetase in Complex with Comp... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6yku | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Neisseria gonorrhoeae Leucyl-tRNA Synthetase in Complex with Compound 11f | ||||||||||||
 Components Components | Leucine--tRNA ligase | ||||||||||||
 Keywords Keywords | LIGASE / protein-inhibitor complex / Rossmann fold / tRNA synthetase | ||||||||||||
| Function / homology |  Function and homology information Function and homology informationleucine-tRNA ligase / leucine-tRNA ligase activity / leucyl-tRNA aminoacylation / aminoacyl-tRNA deacylase activity / ATP binding / cytoplasm / cytosol Similarity search - Function | ||||||||||||
| Biological species |  Neisseria gonorrhoeae (bacteria) Neisseria gonorrhoeae (bacteria) | ||||||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.14 Å MOLECULAR REPLACEMENT / Resolution: 2.14 Å | ||||||||||||
 Authors Authors | Pang, L. / Strelkov, S.V. / Weeks, S.D. | ||||||||||||
| Funding support |  Belgium, 3items Belgium, 3items
| ||||||||||||
 Citation Citation |  Journal: Eur.J.Med.Chem. / Year: 2021 Journal: Eur.J.Med.Chem. / Year: 2021Title: Synthesis and structure-activity studies of novel anhydrohexitol-based Leucyl-tRNA synthetase inhibitors. Authors: De Ruysscher, D. / Pang, L. / Lenders, S.M.G. / Cappoen, D. / Cos, P. / Rozenski, J. / Strelkov, S.V. / Weeks, S.D. / Van Aerschot, A. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6yku.cif.gz 6yku.cif.gz | 353 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6yku.ent.gz pdb6yku.ent.gz | 283.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6yku.json.gz 6yku.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  6yku_validation.pdf.gz 6yku_validation.pdf.gz | 718.9 KB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  6yku_full_validation.pdf.gz 6yku_full_validation.pdf.gz | 723.7 KB | Display | |
| Data in XML |  6yku_validation.xml.gz 6yku_validation.xml.gz | 32.3 KB | Display | |
| Data in CIF |  6yku_validation.cif.gz 6yku_validation.cif.gz | 47.7 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/yk/6yku https://data.pdbj.org/pub/pdb/validation_reports/yk/6yku ftp://data.pdbj.org/pub/pdb/validation_reports/yk/6yku ftp://data.pdbj.org/pub/pdb/validation_reports/yk/6yku | HTTPS FTP |
-Related structure data
| Related structure data |  6ykkC  6yklC  6yknC  6ykoC  6ykqC  6yksC  6yktC  6ykvC  6ykwC  6ykxC  7a0pC  6q89S C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 1 molecules A
| #1: Protein | Mass: 98185.344 Da / Num. of mol.: 1 / Fragment: Leucyl-tRNA Synthetase / Mutation: D454N Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Neisseria gonorrhoeae (bacteria) / Gene: leuS, VT05_02036, WHOO_00006, WHOO_00455 / Plasmid: pETRUK / Production host: Neisseria gonorrhoeae (bacteria) / Gene: leuS, VT05_02036, WHOO_00006, WHOO_00455 / Plasmid: pETRUK / Production host:  References: UniProt: A0A5K1KQ39, UniProt: Q5FAJ3*PLUS, leucine-tRNA ligase |
|---|
-Non-polymers , 5 types, 347 molecules 







| #2: Chemical | ChemComp-ZN / | ||||
|---|---|---|---|---|---|
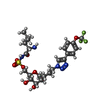
| #3: Chemical | ChemComp-OVB / [( | ||||
| #4: Chemical | | #5: Chemical | ChemComp-MG / | #6: Water | ChemComp-HOH / | |
-Details
| Has ligand of interest | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.26 Å3/Da / Density % sol: 45.61 % / Mosaicity: 0.12 ° |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 8.5 Details: Holo protein at 10 mg/mL in 10 mM Tris pH 7, 100 mM NaCl, 2.5 mM 2-mercaptoethanol was mixed with 0.1 M bis-tris propane pH 8.5, 0.1 M MgCl2, 20% w/v PEG 3350 and a crystal seed stock in a 0. ...Details: Holo protein at 10 mg/mL in 10 mM Tris pH 7, 100 mM NaCl, 2.5 mM 2-mercaptoethanol was mixed with 0.1 M bis-tris propane pH 8.5, 0.1 M MgCl2, 20% w/v PEG 3350 and a crystal seed stock in a 0.75:1.0:0.25 (v/v) ratio. The seed stock was prepared in the same crystallization buffer. Suitable crystals were soaked with 2 mM synthesized compound 11f in an equilvalent precipitant solution supplemented with 22% v/v ethylene glycol. |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SOLEIL SOLEIL  / Beamline: PROXIMA 1 / Wavelength: 0.978566 Å / Beamline: PROXIMA 1 / Wavelength: 0.978566 Å | ||||||||||||||||||||||||||||||
| Detector | Type: DECTRIS EIGER X 16M / Detector: PIXEL / Date: Feb 15, 2019 | ||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.978566 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||
| Reflection | Resolution: 2.1→112.01 Å / Num. obs: 53182 / % possible obs: 99.6 % / Redundancy: 10.1 % / CC1/2: 0.999 / Rmerge(I) obs: 0.096 / Rpim(I) all: 0.031 / Rrim(I) all: 0.101 / Net I/σ(I): 14.3 / Num. measured all: 537780 | ||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 6Q89 Resolution: 2.14→65.498 Å / SU ML: 0.25 / Cross valid method: THROUGHOUT / σ(F): 1.36 / Phase error: 22.77
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 151.97 Å2 / Biso mean: 48.9099 Å2 / Biso min: 21.75 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 2.14→65.498 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj




