| 登録情報 | データベース: PDB / ID: 6va1
|
|---|
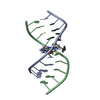
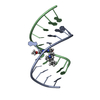
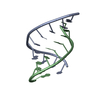
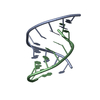
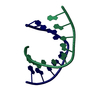
| タイトル | Solution Structure of the Tau pre-mRNA Exon 10 Splicing Regulatory Element |
|---|
 要素 要素 | - RNA (5'-R(*CP*AP*CP*AP*CP*GP*UP*CP*GP*G)-3')
- RNA (5'-R(*CP*CP*GP*GP*CP*AP*GP*UP*GP*UP*G)-3')
|
|---|
 キーワード キーワード | RNA / Adenine bulge / RNA complex / A-form tau RNA hairpin |
|---|
| 機能・相同性 | RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Chen, J.L. / Fountain, M.A. / Disney, M.D. |
|---|
| 資金援助 |  米国, 3件 米国, 3件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | R01-GM097455-07 |  米国 米国 | | National Institutes of Health/National Institute of Neurological Disorders and Stroke (NIH/NINDS) | DP1-NS096898 |  米国 米国 | | National Institutes of Health/National Institute of Neurological Disorders and Stroke (NIH/NINDS) | P01-NS099114 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2020 ジャーナル: J.Am.Chem.Soc. / 年: 2020
タイトル: Design, Optimization, and Study of Small Molecules That Target Tau Pre-mRNA and Affect Splicing.
著者: Chen, J.L. / Zhang, P. / Abe, M. / Aikawa, H. / Zhang, L. / Frank, A.J. / Zembryski, T. / Hubbs, C. / Park, H. / Withka, J. / Steppan, C. / Rogers, L. / Cabral, S. / Pettersson, M. / Wager, T. ...著者: Chen, J.L. / Zhang, P. / Abe, M. / Aikawa, H. / Zhang, L. / Frank, A.J. / Zembryski, T. / Hubbs, C. / Park, H. / Withka, J. / Steppan, C. / Rogers, L. / Cabral, S. / Pettersson, M. / Wager, T.T. / Fountain, M.A. / Rumbaugh, G. / Childs-Disney, J.L. / Disney, M.D. |
|---|
| 履歴 | | 登録 | 2019年12月16日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2020年5月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年5月27日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID |
|---|
| 改定 1.2 | 2024年5月1日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2020
ジャーナル: J.Am.Chem.Soc. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6va1.cif.gz
6va1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6va1.ent.gz
pdb6va1.ent.gz PDB形式
PDB形式 6va1.json.gz
6va1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6va1_validation.pdf.gz
6va1_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6va1_full_validation.pdf.gz
6va1_full_validation.pdf.gz 6va1_validation.xml.gz
6va1_validation.xml.gz 6va1_validation.cif.gz
6va1_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/va/6va1
https://data.pdbj.org/pub/pdb/validation_reports/va/6va1 ftp://data.pdbj.org/pub/pdb/validation_reports/va/6va1
ftp://data.pdbj.org/pub/pdb/validation_reports/va/6va1
 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj





























 NMRPipe
NMRPipe