[English] 日本語
 Yorodumi
Yorodumi- PDB-6ukm: STING C-terminal Domain Complexed with Non-cyclic Dinucleotide Co... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6ukm | ||||||
|---|---|---|---|---|---|---|---|
| Title | STING C-terminal Domain Complexed with Non-cyclic Dinucleotide Compound MSA-2 | ||||||
 Components Components | fusion protein of Ubiquitin-like protein SMT3 and Stimulator of interferon protein c-terminal domain | ||||||
 Keywords Keywords | IMMUNE SYSTEM / AGONIST / STING (STIMULATOR OF INTERFERON GENES) / TRANSMEMBRANE PROTEIN 173 (TMEM173) / IMMUNE SYSTEM-INHIBITOR COMPLEX / IMMUNE SYSTEM-AGONIST COMPLEX | ||||||
| Function / homology |  Function and homology information Function and homology informationSUMO is conjugated to E1 (UBA2:SAE1) / SUMOylation of nuclear envelope proteins / SUMO is transferred from E1 to E2 (UBE2I, UBC9) / SUMO is proteolytically processed / SUMOylation of transcription factors / Postmitotic nuclear pore complex (NPC) reformation / SUMOylation of transcription cofactors / septin ring / SUMOylation of DNA damage response and repair proteins / Transcriptional and post-translational regulation of MITF-M expression and activity ...SUMO is conjugated to E1 (UBA2:SAE1) / SUMOylation of nuclear envelope proteins / SUMO is transferred from E1 to E2 (UBE2I, UBC9) / SUMO is proteolytically processed / SUMOylation of transcription factors / Postmitotic nuclear pore complex (NPC) reformation / SUMOylation of transcription cofactors / septin ring / SUMOylation of DNA damage response and repair proteins / Transcriptional and post-translational regulation of MITF-M expression and activity / SUMOylation of DNA replication proteins / 2',3'-cyclic GMP-AMP binding / SUMOylation of SUMOylation proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / SUMOylation of RNA binding proteins / SUMOylation of chromatin organization proteins / ubiquitin-like protein ligase binding / autophagosome membrane / protein sumoylation / positive regulation of type I interferon production / endoplasmic reticulum-Golgi intermediate compartment membrane / activation of innate immune response / condensed nuclear chromosome / protein tag activity / mitochondrial outer membrane / Golgi membrane / endoplasmic reticulum membrane / perinuclear region of cytoplasm / identical protein binding / nucleus Similarity search - Function | ||||||
| Biological species |   Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.74 Å MOLECULAR REPLACEMENT / Resolution: 1.74 Å | ||||||
 Authors Authors | Lesburg, C.A. | ||||||
 Citation Citation |  Journal: Science / Year: 2020 Journal: Science / Year: 2020Title: An orally available non-nucleotide STING agonist with antitumor activity. Authors: Pan, B.S. / Perera, S.A. / Piesvaux, J.A. / Presland, J.P. / Schroeder, G.K. / Cumming, J.N. / Trotter, B.W. / Altman, M.D. / Buevich, A.V. / Cash, B. / Cemerski, S. / Chang, W. / Chen, Y. / ...Authors: Pan, B.S. / Perera, S.A. / Piesvaux, J.A. / Presland, J.P. / Schroeder, G.K. / Cumming, J.N. / Trotter, B.W. / Altman, M.D. / Buevich, A.V. / Cash, B. / Cemerski, S. / Chang, W. / Chen, Y. / Dandliker, P.J. / Feng, G. / Haidle, A. / Henderson, T. / Jewell, J. / Kariv, I. / Knemeyer, I. / Kopinja, J. / Lacey, B.M. / Laskey, J. / Lesburg, C.A. / Liang, R. / Long, B.J. / Lu, M. / Ma, Y. / Minnihan, E.C. / O'Donnell, G. / Otte, R. / Price, L. / Rakhilina, L. / Sauvagnat, B. / Sharma, S. / Tyagarajan, S. / Woo, H. / Wyss, D.F. / Xu, S. / Bennett, D.J. / Addona, G.H. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6ukm.cif.gz 6ukm.cif.gz | 94.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6ukm.ent.gz pdb6ukm.ent.gz | 68.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6ukm.json.gz 6ukm.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/uk/6ukm https://data.pdbj.org/pub/pdb/validation_reports/uk/6ukm ftp://data.pdbj.org/pub/pdb/validation_reports/uk/6ukm ftp://data.pdbj.org/pub/pdb/validation_reports/uk/6ukm | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6ukuC  6ukvC  6ukwC  6ukxC  6ukyC  6ukzC  6ul0C  4ksyS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
| ||||||||
| Components on special symmetry positions |
|
- Components
Components
| #1: Protein | Mass: 34456.527 Da / Num. of mol.: 1 / Mutation: G230A,R293Q Source method: isolated from a genetically manipulated source Source: (gene. exp.)   Homo sapiens (human) Homo sapiens (human)Strain: ATCC 204508 / S288c Gene: SMT3, YDR510W, D9719.15, STING, LOC340061, hCG_1782396 Plasmid: pET47b / Production host:  |
|---|---|
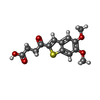
| #2: Chemical | ChemComp-QAD / |
| #3: Water | ChemComp-HOH / |
| Has ligand of interest | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.54 Å3/Da / Density % sol: 20.05 % / Mosaicity: 0.45 ° |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion / pH: 8.5 / Details: 25% PEG 6000, 100 mM Tris, 200 mM NaCl, 2 mM DTT |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 17-ID / Wavelength: 1 Å / Beamline: 17-ID / Wavelength: 1 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Mar 9, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 1.738→47.9 Å / Num. obs: 19712 / % possible obs: 91.3 % / Redundancy: 3.3 % / Biso Wilson estimate: 30.15 Å2 / CC1/2: 0.997 / Rpim(I) all: 0.039 / Rrim(I) all: 0.073 / Net I/σ(I): 12.1 |
| Reflection shell | Resolution: 1.738→1.744 Å / Redundancy: 3.4 % / Mean I/σ(I) obs: 2.4 / Num. measured all: 10069 / Num. unique obs: 3031 / CC1/2: 0.797 / Rpim(I) all: 0.394 / Rrim(I) all: 0.73 / Net I/σ(I) obs: 2.1 / % possible all: 94.5 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: pdbid 4KSY Resolution: 1.74→47.9 Å / Cor.coef. Fo:Fc: 0.9534 / Cor.coef. Fo:Fc free: 0.9514 / SU R Cruickshank DPI: 0.111 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.112 / SU Rfree Blow DPI: 0.098 / SU Rfree Cruickshank DPI: 0.098
| ||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 158.14 Å2 / Biso mean: 38.45 Å2 / Biso min: 11.16 Å2
| ||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.227 Å | ||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.74→47.9 Å
| ||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.74→1.83 Å / Rfactor Rfree error: 0 / Total num. of bins used: 10
| ||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Origin x: 41.0684 Å / Origin y: 5.5377 Å / Origin z: -18.9822 Å
| ||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group | Selection details: { A|* } |
 Movie
Movie Controller
Controller












 PDBj
PDBj