+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6tt8 | ||||||
|---|---|---|---|---|---|---|---|
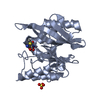
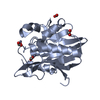
| タイトル | Haddock model of NDM-1/morin complex | ||||||
 要素 要素 | Metallo beta lactamase NDM-1 | ||||||
 キーワード キーワード | METAL BINDING PROTEIN / complex / inhibitor / morin / New-Delhi Metallo-beta-lactamase-1 / Haddock / antimicrobial resistance | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Riviere, G. / Oueslati, S. / Gayral, M. / Crechet, J.B. / Nhiri, N. / Jacquet, E. / Cintrat, J.C. / Giraud, F. / van Heijenoort, C. / Lescop, E. ...Riviere, G. / Oueslati, S. / Gayral, M. / Crechet, J.B. / Nhiri, N. / Jacquet, E. / Cintrat, J.C. / Giraud, F. / van Heijenoort, C. / Lescop, E. / Pethe, S. / Iorga, B.I. / Naas, T. / Guittet, E. / Morellet, N. | ||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| ||||||
 引用 引用 |  ジャーナル: Acs Omega / 年: 2020 ジャーナル: Acs Omega / 年: 2020タイトル: NMR Characterization of the Influence of Zinc(II) Ions on the Structural and Dynamic Behavior of the New Delhi Metallo-beta-Lactamase-1 and on the Binding with Flavonols as Inhibitors. 著者: Riviere, G. / Oueslati, S. / Gayral, M. / Crechet, J.B. / Nhiri, N. / Jacquet, E. / Cintrat, J.C. / Giraud, F. / van Heijenoort, C. / Lescop, E. / Pethe, S. / Iorga, B.I. / Naas, T. / Guittet, E. / Morellet, N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6tt8.cif.gz 6tt8.cif.gz | 3.5 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6tt8.ent.gz pdb6tt8.ent.gz | 3 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6tt8.json.gz 6tt8.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6tt8_validation.pdf.gz 6tt8_validation.pdf.gz | 485.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6tt8_full_validation.pdf.gz 6tt8_full_validation.pdf.gz | 737.8 KB | 表示 | |
| XML形式データ |  6tt8_validation.xml.gz 6tt8_validation.xml.gz | 256.2 KB | 表示 | |
| CIF形式データ |  6tt8_validation.cif.gz 6tt8_validation.cif.gz | 307.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tt/6tt8 https://data.pdbj.org/pub/pdb/validation_reports/tt/6tt8 ftp://data.pdbj.org/pub/pdb/validation_reports/tt/6tt8 ftp://data.pdbj.org/pub/pdb/validation_reports/tt/6tt8 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ttaC  6ttcC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 23974.957 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌)遺伝子: blaNDM-1, bla NDM-1, blaNDM-1_1, blaNDM-1_2, blaNDM-1_3, blaNDM1, NDM-1, BANRA_05884, C3483_29595, C7V41_28630, D647_p47098, EC13450_007, FAQ72_26810, FAQ97_27095, FAS39_27275, NCTC13443_ ...遺伝子: blaNDM-1, bla NDM-1, blaNDM-1_1, blaNDM-1_2, blaNDM-1_3, blaNDM1, NDM-1, BANRA_05884, C3483_29595, C7V41_28630, D647_p47098, EC13450_007, FAQ72_26810, FAQ97_27095, FAS39_27275, NCTC13443_00040, p2146_00143, pCRE380_21, PMK1_ndm00067, PMK1_ndm00076, PMK1_ndm00085, pN11x00042NDM_090, pNDM-SX04_5, pNDM10469_138, SAMEA3531848_05178, TR3_031, TR4_031 発現宿主:  | ||||
|---|---|---|---|---|---|
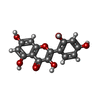
| #2: 化合物 | | #3: 化合物 | ChemComp-MRI / | 研究の焦点であるリガンドがあるか | Y | |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | タイプ: solution 内容: 320 uM [U-13C; U-15N] NDM-1, 1280 uM morin, 0.1 mM Bis-tris, 150 mM sodium chloride, 2.5 % DMSO, 90% H2O/10% D2O Label: 15N, 13C sample / 溶媒系: 90% H2O/10% D2O | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||
| 試料状態 | イオン強度: 150 mM / Label: condition_1 / pH: 7 / PH err: 0.05 / 圧: 1 atm / 温度: 293 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 4 | |||||||||||||||
| 代表構造 | 選択基準: closest to the average | |||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 2800 / 登録したコンフォーマーの数: 56 |
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj




 HSQC
HSQC