+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6qlf | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
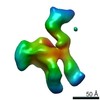
| Title | Structure of inner kinetochore CCAN complex with mask1 | ||||||||||||||||||||||||||||||||||||||||||
 Components Components | (Inner kinetochore subunit ...) x 8 | ||||||||||||||||||||||||||||||||||||||||||
 Keywords Keywords | DNA BINDING PROTEIN / inner kinetochore / CCAN | ||||||||||||||||||||||||||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of meiotic DNA double-strand break formation involved in reciprocal meiotic recombination / COMA complex / maintenance of meiotic sister chromatid cohesion / : / Mis6-Sim4 complex / meiotic sister chromatid segregation / establishment of meiotic sister chromatid cohesion / ascospore formation / attachment of spindle microtubules to kinetochore / CENP-A containing chromatin assembly ...negative regulation of meiotic DNA double-strand break formation involved in reciprocal meiotic recombination / COMA complex / maintenance of meiotic sister chromatid cohesion / : / Mis6-Sim4 complex / meiotic sister chromatid segregation / establishment of meiotic sister chromatid cohesion / ascospore formation / attachment of spindle microtubules to kinetochore / CENP-A containing chromatin assembly / protein localization to chromosome, centromeric region / kinetochore assembly / outer kinetochore / establishment of mitotic sister chromatid cohesion / protein localization to kinetochore / spindle pole body / mitotic spindle assembly checkpoint signaling / meiotic cell cycle / chromosome segregation / kinetochore / cell division / structural molecule activity / nucleus / cytoplasm Similarity search - Function | ||||||||||||||||||||||||||||||||||||||||||
| Biological species |  | ||||||||||||||||||||||||||||||||||||||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 3.45 Å | ||||||||||||||||||||||||||||||||||||||||||
 Authors Authors | Yan, K. / Yang, J. / Zhang, Z. / McLaughlin, S.H. / Chang, L. / Fasci, D. / Heck, A.J.R. / Barford, D. | ||||||||||||||||||||||||||||||||||||||||||
| Funding support |  United Kingdom, 3items United Kingdom, 3items
| ||||||||||||||||||||||||||||||||||||||||||
 Citation Citation |  Journal: Nature / Year: 2019 Journal: Nature / Year: 2019Title: Structure of the inner kinetochore CCAN complex assembled onto a centromeric nucleosome. Authors: Kaige Yan / Jing Yang / Ziguo Zhang / Stephen H McLaughlin / Leifu Chang / Domenico Fasci / Ann E Ehrenhofer-Murray / Albert J R Heck / David Barford /     Abstract: In eukaryotes, accurate chromosome segregation in mitosis and meiosis maintains genome stability and prevents aneuploidy. Kinetochores are large protein complexes that, by assembling onto specialized ...In eukaryotes, accurate chromosome segregation in mitosis and meiosis maintains genome stability and prevents aneuploidy. Kinetochores are large protein complexes that, by assembling onto specialized Cenp-A nucleosomes, function to connect centromeric chromatin to microtubules of the mitotic spindle. Whereas the centromeres of vertebrate chromosomes comprise millions of DNA base pairs and attach to multiple microtubules, the simple point centromeres of budding yeast are connected to individual microtubules. All 16 budding yeast chromosomes assemble complete kinetochores using a single Cenp-A nucleosome (Cenp-A), each of which is perfectly centred on its cognate centromere. The inner and outer kinetochore modules are responsible for interacting with centromeric chromatin and microtubules, respectively. Here we describe the cryo-electron microscopy structure of the Saccharomyces cerevisiae inner kinetochore module, the constitutive centromere associated network (CCAN) complex, assembled onto a Cenp-A nucleosome (CCAN-Cenp-A). The structure explains the interdependency of the constituent subcomplexes of CCAN and shows how the Y-shaped opening of CCAN accommodates Cenp-A to enable specific CCAN subunits to contact the nucleosomal DNA and histone subunits. Interactions with the unwrapped DNA duplex at the two termini of Cenp-A are mediated predominantly by a DNA-binding groove in the Cenp-L-Cenp-N subcomplex. Disruption of these interactions impairs assembly of CCAN onto Cenp-A. Our data indicate a mechanism of Cenp-A nucleosome recognition by CCAN and how CCAN acts as a platform for assembly of the outer kinetochore to link centromeres to the mitotic spindle for chromosome segregation. | ||||||||||||||||||||||||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6qlf.cif.gz 6qlf.cif.gz | 334 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6qlf.ent.gz pdb6qlf.ent.gz | 254.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6qlf.json.gz 6qlf.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ql/6qlf https://data.pdbj.org/pub/pdb/validation_reports/ql/6qlf ftp://data.pdbj.org/pub/pdb/validation_reports/ql/6qlf ftp://data.pdbj.org/pub/pdb/validation_reports/ql/6qlf | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4581MC  4579C  4580C  4971C  6qldC  6qleC M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
-Inner kinetochore subunit ... , 8 types, 8 molecules LNOPQUYZ
| #1: Protein | Mass: 28093.223 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: IML3, MCM19, YBR107C, YBR0836 / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: P38265 Trichoplusia ni (cabbage looper) / References: UniProt: P38265 |
|---|---|
| #2: Protein | Mass: 53538.566 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: CHL4, CTF17, MCM17, YDR254W, YD9320A.04 / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: P38907 Trichoplusia ni (cabbage looper) / References: UniProt: P38907 |
| #3: Protein | Mass: 43028.879 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: MCM21, CTF5, YDR318W / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: Q06675 Trichoplusia ni (cabbage looper) / References: UniProt: Q06675 |
| #4: Protein | Mass: 42841.113 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: CTF19, MCM18, YPL018W, LPB13W / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: Q02732 Trichoplusia ni (cabbage looper) / References: UniProt: Q02732 |
| #5: Protein | Mass: 47427.246 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: OKP1, YGR179C / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: P53298 Trichoplusia ni (cabbage looper) / References: UniProt: P53298 |
| #6: Protein | Mass: 37081.246 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: AME1, ARP100, YBR211C, YBR1458 / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: P38313 Trichoplusia ni (cabbage looper) / References: UniProt: P38313 |
| #7: Protein | Mass: 27006.451 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: NKP1, YDR383C / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: Q12493 Trichoplusia ni (cabbage looper) / References: UniProt: Q12493 |
| #8: Protein | Mass: 17877.033 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Gene: NKP2, YLR315W / Cell line (production host): High Five / Production host:  Trichoplusia ni (cabbage looper) / References: UniProt: Q06162 Trichoplusia ni (cabbage looper) / References: UniProt: Q06162 |
-Details
| Has protein modification | N |
|---|
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component | Name: Inner kinetochore CCAN complex / Type: COMPLEX / Entity ID: all / Source: RECOMBINANT |
|---|---|
| Source (natural) | Organism:  |
| Source (recombinant) | Organism:  Trichoplusia ni (cabbage looper) Trichoplusia ni (cabbage looper) |
| Buffer solution | pH: 8 |
| Specimen | Conc.: 1 mg/ml / Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES |
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: FEI TITAN KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD |
| Image recording | Electron dose: 32 e/Å2 / Detector mode: COUNTING / Film or detector model: FEI FALCON III (4k x 4k) |
- Processing
Processing
| Software | Name: PHENIX / Version: 1.14_3260: / Classification: refinement |
|---|---|
| EM software | Name: PHENIX / Category: model refinement |
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION |
| 3D reconstruction | Resolution: 3.45 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 465029 / Symmetry type: POINT |
 Movie
Movie Controller
Controller










 PDBj
PDBj