| 登録情報 | データベース: PDB / ID: 6qdj
|
|---|
| タイトル | Molecular features of the UNC-45 chaperone critical for binding and folding muscle myosin |
|---|
 要素 要素 | Myosin-4 |
|---|
 キーワード キーワード | MOTOR PROTEIN / MYOSIN / MHC-B / UNC-54 |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
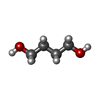
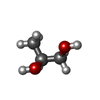
egg-laying behavior / striated muscle myosin thick filament / skeletal muscle myosin thick filament assembly / A band / muscle myosin complex / myosin filament / locomotion / myosin II complex / structural constituent of muscle / sarcomere organization ...egg-laying behavior / striated muscle myosin thick filament / skeletal muscle myosin thick filament assembly / A band / muscle myosin complex / myosin filament / locomotion / myosin II complex / structural constituent of muscle / sarcomere organization / microfilament motor activity / muscle contraction / actin filament binding / ATP binding / cytoplasm類似検索 - 分子機能 Methane Monooxygenase Hydroxylase; Chain G, domain 1 - #60 / DNA repair protein XRCC4-like, C-terminal / Myosin tail / Myosin tail / Myosin N-terminal SH3-like domain / Myosin S1 fragment, N-terminal / Myosin, N-terminal, SH3-like / Myosin N-terminal SH3-like domain profile. / Myosin head, motor domain / Myosin head (motor domain) ...Methane Monooxygenase Hydroxylase; Chain G, domain 1 - #60 / DNA repair protein XRCC4-like, C-terminal / Myosin tail / Myosin tail / Myosin N-terminal SH3-like domain / Myosin S1 fragment, N-terminal / Myosin, N-terminal, SH3-like / Myosin N-terminal SH3-like domain profile. / Myosin head, motor domain / Myosin head (motor domain) / Myosin motor domain profile. / Myosin. Large ATPases. / Kinesin motor domain superfamily / Methane Monooxygenase Hydroxylase; Chain G, domain 1 / Up-down Bundle / P-loop containing nucleoside triphosphate hydrolase / Mainly Alpha類似検索 - ドメイン・相同性 ADENOSINE-5'-DIPHOSPHATE / 1,4-BUTANEDIOL / HEXANE-1,6-DIOL / 2,5,8,11,14,17-HEXAOXANONADECAN-19-OL / S-1,2-PROPANEDIOL / Myosin-4類似検索 - 構成要素 |
|---|
| 生物種 |   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.884 Å 分子置換 / 解像度: 1.884 Å |
|---|
 データ登録者 データ登録者 | Meinhart, A. / Clausen, T. / Arnese, R. |
|---|
| 資金援助 |  オーストリア, 1件 オーストリア, 1件 | 組織 | 認可番号 | 国 |
|---|
| Austrian Science Fund | P 22570 |  オーストリア オーストリア |
|
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019
タイトル: Molecular features of the UNC-45 chaperone critical for binding and folding muscle myosin.
著者: Hellerschmied, D. / Lehner, A. / Franicevic, N. / Arnese, R. / Johnson, C. / Vogel, A. / Meinhart, A. / Kurzbauer, R. / Deszcz, L. / Gazda, L. / Geeves, M. / Clausen, T. |
|---|
| 履歴 | | 登録 | 2019年1月2日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2019年10月30日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年1月24日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.884 Å
分子置換 / 解像度: 1.884 Å  データ登録者
データ登録者 オーストリア, 1件
オーストリア, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2019
ジャーナル: Nat Commun / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6qdj.cif.gz
6qdj.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6qdj.ent.gz
pdb6qdj.ent.gz PDB形式
PDB形式 6qdj.json.gz
6qdj.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6qdj_validation.pdf.gz
6qdj_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6qdj_full_validation.pdf.gz
6qdj_full_validation.pdf.gz 6qdj_validation.xml.gz
6qdj_validation.xml.gz 6qdj_validation.cif.gz
6qdj_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/qd/6qdj
https://data.pdbj.org/pub/pdb/validation_reports/qd/6qdj ftp://data.pdbj.org/pub/pdb/validation_reports/qd/6qdj
ftp://data.pdbj.org/pub/pdb/validation_reports/qd/6qdj リンク
リンク 集合体
集合体
 要素
要素
 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P02566
Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P02566









 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  PETRA III, EMBL c/o DESY
PETRA III, EMBL c/o DESY  / ビームライン: P13 (MX1) / 波長: 0.9763 Å
/ ビームライン: P13 (MX1) / 波長: 0.9763 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj














