| #1: タンパク質 | Beta-D-glucuronidase / Beta-glucuronidase / NLS-GFP-GUS
分子量: 68804.000 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌)遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, ...遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, BK292_18725, BOH76_23450, BON63_17270, BON69_09035, BON71_25965, BUE81_02440, BvCms2454_03287, BvCmsHHP019_03955, BvCmsKKP061_00687, BvCmsKSP011_01150, BvCmsKSP024_03693, BvCmsKSP026_03508, BvCmsKSP045_00685, BvCmsKSP067_00118, BvCmsNSP047_02620, BvCmsSINP012_02859, BvCmsSIP019_00145, BVL39_19890, BW690_15740, BZL31_05290, C4J69_24575, C5N07_00485, C7235_11830, C9200_15785, C9E25_02740, C9Z12_02165, CA593_19915, CI694_12700, COD46_02605, CV83915_04776, D2185_18760, D3821_14300, D3Y67_19800, D9D20_10680, D9I11_03210, D9K48_06425, DBQ99_12860, DNQ41_12380, DXT69_06115, DXT71_07500, E2119_03270, E5P28_03750, E5S47_08220, EAI46_18055, EAI52_05705, EC3234A_33c01180, EC3426_02616, EEP23_13620, EL75_2040, EL79_2080, EL80_2107, ELT20_01525, EPS71_15340, ERS085365_00730, ERS085416_00883, ERS139211_01060, ERS150873_00637, EXX24_04480, EXX78_14190, EYD11_10940, EYY78_00120, FAX15_15905, FV293_04650, NCTC11181_05093, NCTC13462_05935, NCTC8500_02703, NCTC9045_02849, NCTC9062_00385, NCTC9706_03872, PU06_11555, RG28_14030, RK56_009440, SAMEA3472044_02684, SAMEA3472047_01270, SAMEA3472080_01417, SAMEA3484427_00776, SAMEA3484429_00887, SAMEA3752553_00640, SAMEA3752559_02259, SAMEA3753097_02320, SK85_01835, WR15_14365発現宿主 Escherichia coli (大腸菌)遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, ...遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, BK292_18725, BOH76_23450, BON63_17270, BON69_09035, BON71_25965, BUE81_02440, BvCms2454_03287, BvCmsHHP019_03955, BvCmsKKP061_00687, BvCmsKSP011_01150, BvCmsKSP024_03693, BvCmsKSP026_03508, BvCmsKSP045_00685, BvCmsKSP067_00118, BvCmsNSP047_02620, BvCmsSINP012_02859, BvCmsSIP019_00145, BVL39_19890, BW690_15740, BZL31_05290, C4J69_24575, C5N07_00485, C7235_11830, C9200_15785, C9E25_02740, C9Z12_02165, CA593_19915, CI694_12700, COD46_02605, CV83915_04776, D2185_18760, D3821_14300, D3Y67_19800, D9D20_10680, D9I11_03210, D9K48_06425, DBQ99_12860, DNQ41_12380, DXT69_06115, DXT71_07500, E2119_03270, E5P28_03750, E5S47_08220, EAI46_18055, EAI52_05705, EC3234A_33c01180, EC3426_02616, EEP23_13620, EL75_2040, EL79_2080, EL80_2107, ELT20_01525, EPS71_15340, ERS085365_00730, ERS085416_00883, ERS139211_01060, ERS150873_00637, EXX24_04480, EXX78_14190, EYD11_10940, EYY78_00120, FAX15_15905, FV293_04650, NCTC11181_05093, NCTC13462_05935, NCTC8500_02703, NCTC9045_02849, NCTC9062_00385, NCTC9706_03872, PU06_11555, RG28_14030, RK56_009440, SAMEA3472044_02684, SAMEA3472047_01270, SAMEA3472080_01417, SAMEA3484427_00776, SAMEA3484429_00887, SAMEA3752553_00640, SAMEA3752559_02259, SAMEA3753097_02320, SK85_01835, WR15_14365発現宿主:   Escherichia coli (大腸菌)参照 Escherichia coli (大腸菌)参照: UniProt: W8SYR0, UniProt: P05804*PLUS, beta-glucuronidase       |
|---|
| #2: タンパク質 | Beta-D-glucuronidase / Beta-glucuronidase / NLS-GFP-GUS
分子量: 68521.727 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌)遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, ...遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, BK292_18725, BOH76_23450, BON63_17270, BON69_09035, BON71_25965, BUE81_02440, BvCms2454_03287, BvCmsHHP019_03955, BvCmsKKP061_00687, BvCmsKSP011_01150, BvCmsKSP024_03693, BvCmsKSP026_03508, BvCmsKSP045_00685, BvCmsKSP067_00118, BvCmsNSP047_02620, BvCmsSINP012_02859, BvCmsSIP019_00145, BVL39_19890, BW690_15740, BZL31_05290, C4J69_24575, C5N07_00485, C7235_11830, C9200_15785, C9E25_02740, C9Z12_02165, CA593_19915, CI694_12700, COD46_02605, CV83915_04776, D2185_18760, D3821_14300, D3Y67_19800, D9D20_10680, D9I11_03210, D9K48_06425, DBQ99_12860, DNQ41_12380, DXT69_06115, DXT71_07500, E2119_03270, E5P28_03750, E5S47_08220, EAI46_18055, EAI52_05705, EC3234A_33c01180, EC3426_02616, EEP23_13620, EL75_2040, EL79_2080, EL80_2107, ELT20_01525, EPS71_15340, ERS085365_00730, ERS085416_00883, ERS139211_01060, ERS150873_00637, EXX24_04480, EXX78_14190, EYD11_10940, EYY78_00120, FAX15_15905, FV293_04650, NCTC11181_05093, NCTC13462_05935, NCTC8500_02703, NCTC9045_02849, NCTC9062_00385, NCTC9706_03872, PU06_11555, RG28_14030, RK56_009440, SAMEA3472044_02684, SAMEA3472047_01270, SAMEA3472080_01417, SAMEA3484427_00776, SAMEA3484429_00887, SAMEA3752553_00640, SAMEA3752559_02259, SAMEA3753097_02320, SK85_01835, WR15_14365発現宿主 Escherichia coli (大腸菌)遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, ...遺伝子: uidA, A9R57_02750, ACN68_26240, ACN81_11215, ACU57_22480, AM270_03870, AM464_25945, AUQ13_03930, BANRA_00079, BANRA_00563, BANRA_02251, BB545_08625, BET08_08370, BHS87_09195, BJJ90_13245, BK292_18725, BOH76_23450, BON63_17270, BON69_09035, BON71_25965, BUE81_02440, BvCms2454_03287, BvCmsHHP019_03955, BvCmsKKP061_00687, BvCmsKSP011_01150, BvCmsKSP024_03693, BvCmsKSP026_03508, BvCmsKSP045_00685, BvCmsKSP067_00118, BvCmsNSP047_02620, BvCmsSINP012_02859, BvCmsSIP019_00145, BVL39_19890, BW690_15740, BZL31_05290, C4J69_24575, C5N07_00485, C7235_11830, C9200_15785, C9E25_02740, C9Z12_02165, CA593_19915, CI694_12700, COD46_02605, CV83915_04776, D2185_18760, D3821_14300, D3Y67_19800, D9D20_10680, D9I11_03210, D9K48_06425, DBQ99_12860, DNQ41_12380, DXT69_06115, DXT71_07500, E2119_03270, E5P28_03750, E5S47_08220, EAI46_18055, EAI52_05705, EC3234A_33c01180, EC3426_02616, EEP23_13620, EL75_2040, EL79_2080, EL80_2107, ELT20_01525, EPS71_15340, ERS085365_00730, ERS085416_00883, ERS139211_01060, ERS150873_00637, EXX24_04480, EXX78_14190, EYD11_10940, EYY78_00120, FAX15_15905, FV293_04650, NCTC11181_05093, NCTC13462_05935, NCTC8500_02703, NCTC9045_02849, NCTC9062_00385, NCTC9706_03872, PU06_11555, RG28_14030, RK56_009440, SAMEA3472044_02684, SAMEA3472047_01270, SAMEA3472080_01417, SAMEA3484427_00776, SAMEA3484429_00887, SAMEA3752553_00640, SAMEA3752559_02259, SAMEA3753097_02320, SK85_01835, WR15_14365発現宿主:   Escherichia coli (大腸菌)参照 Escherichia coli (大腸菌)参照: UniProt: W8SYR0, UniProt: P05804*PLUS, beta-glucuronidase      |
|---|
| #3: 化合物 | ChemComp-CKX / (2~{S},3~{S},4~{R},5~{R})-4,5-bis(oxidanyl)-2-propyl-piperidine-3-carboxylic acid
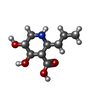  分子量 分子量: 203.236 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C 9H 17NO 4 / タイプ: SUBJECT OF INVESTIGATION | #4: 水 | ChemComp-HOH / water
 分子量 分子量: 18.015 Da / 分子数: 65 / 由来タイプ: 天然 / 式: H 2O |
|---|
| 研究の焦点であるリガンドがあるか | Y |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.617 Å
分子置換 / 解像度: 2.617 Å  データ登録者
データ登録者 台湾, 1件
台湾, 1件  引用
引用 ジャーナル: Commun Biol / 年: 2021
ジャーナル: Commun Biol / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6lej.cif.gz
6lej.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6lej.ent.gz
pdb6lej.ent.gz PDB形式
PDB形式 6lej.json.gz
6lej.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6lej_validation.pdf.gz
6lej_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6lej_full_validation.pdf.gz
6lej_full_validation.pdf.gz 6lej_validation.xml.gz
6lej_validation.xml.gz 6lej_validation.cif.gz
6lej_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/le/6lej
https://data.pdbj.org/pub/pdb/validation_reports/le/6lej ftp://data.pdbj.org/pub/pdb/validation_reports/le/6lej
ftp://data.pdbj.org/pub/pdb/validation_reports/le/6lej リンク
リンク 集合体
集合体

 要素
要素



 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSRRC
NSRRC  / ビームライン: TPS 05A / 波長: 1 Å
/ ビームライン: TPS 05A / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj




