| 登録情報 | データベース: PDB / ID: 6k2k
|
|---|
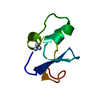
| タイトル | Solution structure of MUL1-RING domain |
|---|
 要素 要素 | Mitochondrial ubiquitin ligase activator of NFKB 1 |
|---|
 キーワード キーワード | STRUCTURAL PROTEIN / Solution structure / E3 ubiquitin ligase |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of mitochondrial outer membrane permeabilization involved in apoptotic signaling pathway / negative regulation of chemokine (C-C motif) ligand 5 production / negative regulation of defense response to virus by host / negative regulation of mitochondrial fusion / mitochondrion localization / positive regulation of dendrite extension / positive regulation of protein sumoylation / positive regulation of type 2 mitophagy / mitochondrial fission / regulation of mitochondrion organization ...regulation of mitochondrial outer membrane permeabilization involved in apoptotic signaling pathway / negative regulation of chemokine (C-C motif) ligand 5 production / negative regulation of defense response to virus by host / negative regulation of mitochondrial fusion / mitochondrion localization / positive regulation of dendrite extension / positive regulation of protein sumoylation / positive regulation of type 2 mitophagy / mitochondrial fission / regulation of mitochondrion organization / SUMO transferase activity / negative regulation of type I interferon-mediated signaling pathway / positive regulation of mitochondrial fission / cellular response to exogenous dsRNA / protein sumoylation / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / negative regulation of innate immune response / regulation of mitochondrial membrane potential / protein destabilization / RING-type E3 ubiquitin transferase / protein polyubiquitination / ubiquitin-protein transferase activity / p53 binding / ubiquitin protein ligase activity / KEAP1-NFE2L2 pathway / peroxisome / Neddylation / mitochondrial outer membrane / positive regulation of canonical NF-kappaB signal transduction / Ub-specific processing proteases / protein stabilization / protein ubiquitination / axon / neuronal cell body / apoptotic process / ubiquitin protein ligase binding / mitochondrion / zinc ion binding / identical protein binding / membrane類似検索 - 分子機能 E3 Ubiquitin ligase MUL1-like / E3 Ubiquitin ligase / : / Zinc finger, C3HC4 type (RING finger) / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / Zinc finger RING-type profile. / Zinc finger, RING-type / Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Mitochondrial ubiquitin ligase activator of NFKB 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Lee, M.S. / Lee, M.K. / Ryu, K.S. / Chi, S.W. |
|---|
 引用 引用 |  ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2019 ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2019
タイトル: Solution structure of MUL1-RING domain and its interaction with p53 transactivation domain.
著者: Lee, M.S. / Lee, S.O. / Lee, M.K. / Yi, G.S. / Lee, C.K. / Ryu, K.S. / Chi, S.W. |
|---|
| 履歴 | | 登録 | 2019年5月14日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2019年7月10日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年7月31日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Database references / Other / カテゴリ: database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.3 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2019
ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6k2k.cif.gz
6k2k.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6k2k.ent.gz
pdb6k2k.ent.gz PDB形式
PDB形式 6k2k.json.gz
6k2k.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6k2k_validation.pdf.gz
6k2k_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6k2k_full_validation.pdf.gz
6k2k_full_validation.pdf.gz 6k2k_validation.xml.gz
6k2k_validation.xml.gz 6k2k_validation.cif.gz
6k2k_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/k2/6k2k
https://data.pdbj.org/pub/pdb/validation_reports/k2/6k2k ftp://data.pdbj.org/pub/pdb/validation_reports/k2/6k2k
ftp://data.pdbj.org/pub/pdb/validation_reports/k2/6k2k リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: MUL1, C1orf166, GIDE, MAPL, MULAN, RNF218 / プラスミド: pGEX-4T3 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: MUL1, C1orf166, GIDE, MAPL, MULAN, RNF218 / プラスミド: pGEX-4T3 / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


 HSQC
HSQC