[English] 日本語
 Yorodumi
Yorodumi- PDB-6hc5: The structure of the G. violaceus guanidine II riboswitch P2 stem... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6hc5 | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
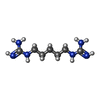
| Title | The structure of the G. violaceus guanidine II riboswitch P2 stem-loop with audouine | ||||||||||||||||||||||||||||
 Components Components | RNA (5'-R(* Keywords KeywordsRNA / guanidine II riboswitch / stem-loop / tetra loop / dimer | Function / homology | 1-(5-carbamimidamidopentyl)guanidine / RNA / RNA (> 10) |  Function and homology information Function and homology informationBiological species |  Gloeobacter violaceus (bacteria) Gloeobacter violaceus (bacteria)Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / Resolution: 1.413 Å SYNCHROTRON / Resolution: 1.413 Å  Authors AuthorsHuang, L. / Lilley, D.M.J. | Funding support | |  United Kingdom, 1items United Kingdom, 1items
 Citation Citation Journal: Rna / Year: 2019 Journal: Rna / Year: 2019Title: Structure-guided design of a high-affinity ligand for a riboswitch. Authors: Huang, L. / Wang, J. / Wilson, T.J. / Lilley, D.M.J. History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6hc5.cif.gz 6hc5.cif.gz | 33.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6hc5.ent.gz pdb6hc5.ent.gz | 23.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6hc5.json.gz 6hc5.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/hc/6hc5 https://data.pdbj.org/pub/pdb/validation_reports/hc/6hc5 ftp://data.pdbj.org/pub/pdb/validation_reports/hc/6hc5 ftp://data.pdbj.org/pub/pdb/validation_reports/hc/6hc5 | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 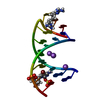
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||
| Components on special symmetry positions |
|
- Components
Components
| #1: RNA chain | Mass: 5880.434 Da / Num. of mol.: 1 / Source method: obtained synthetically / Source: (synth.)  Gloeobacter violaceus (bacteria) Gloeobacter violaceus (bacteria) | ||||
|---|---|---|---|---|---|
| #2: Chemical | ChemComp-SO4 / | ||||
| #3: Chemical | | #4: Chemical | ChemComp-FXT / | #5: Water | ChemComp-HOH / | |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.4 Å3/Da / Density % sol: 63.83 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 5 Details: 0.01 M Magnesium Acetate, 0.05 M MES pH 5.0, 2.5 M Ammonium Sulfate |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Diamond Diamond  / Beamline: I04 / Wavelength: 0.9196 Å / Beamline: I04 / Wavelength: 0.9196 Å |
| Detector | Type: DECTRIS PILATUS3 6M / Detector: PIXEL / Date: Nov 29, 2017 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9196 Å / Relative weight: 1 |
| Reflection | Resolution: 1.413→27.913 Å / Num. obs: 28892 / % possible obs: 98 % / Observed criterion σ(I): 0.8 / Redundancy: 6.3 % / CC1/2: 1 / Rmerge(I) obs: 0.047 / Rpim(I) all: 0.021 / Net I/σ(I): 18.1 |
| Reflection shell | Resolution: 1.413→1.437 Å / Redundancy: 6.5 % / Rmerge(I) obs: 1.92 / Num. unique obs: 775 / CC1/2: 0.428 / Rpim(I) all: 0.813 / % possible all: 100 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Resolution: 1.413→27.913 Å / SU ML: 0.24 / Cross valid method: THROUGHOUT / σ(F): 1.34 / Phase error: 24.1
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.413→27.913 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller







 PDBj
PDBj