| 登録情報 | データベース: PDB / ID: 6gft
|
|---|
| タイトル | Antinociceptive evaluation of cyriotoxin-1a, the first toxin purified from Cyriopagopus schioedtei spider venom |
|---|
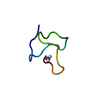
 要素 要素 | cyriotoxin-1a |
|---|
 キーワード キーワード | TOXIN / Toxine / Spider Venom / Cyriopagopus schioedtei |
|---|
| 機能・相同性 | Huwentoxin, conserved site-1 / Huwentoxin-1 family signature. / Huwentoxin-1 family / Ion channel inhibitory toxin / ion channel inhibitor activity / sodium channel regulator activity / toxin activity / extracellular region / Mu-theraphotoxin-Os1a 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Cyriopagopus schioedtei (クモ) Cyriopagopus schioedtei (クモ) |
|---|
| 手法 | 溶液NMR / molecular dynamics |
|---|
 データ登録者 データ登録者 | Kurz, M. |
|---|
| 資金援助 |  フランス, 1件 フランス, 1件 | 組織 | 認可番号 | 国 |
|---|
| French National Research Agency | 153114 |  フランス フランス |
|
|---|
 引用 引用 |  ジャーナル: Br.J.Pharmacol. / 年: 2019 ジャーナル: Br.J.Pharmacol. / 年: 2019
タイトル: From identification to functional characterization of cyriotoxin-1a, an antinociceptive toxin from the spider Cyriopagopus schioedtei.
著者: Goncalves, T.C. / Benoit, E. / Kurz, M. / Lucarain, L. / Fouconnier, S. / Combemale, S. / Jaquillard, L. / Schombert, B. / Chambard, J.M. / Boukaiba, R. / Hessler, G. / Bohme, A. / Bialy, L. ...著者: Goncalves, T.C. / Benoit, E. / Kurz, M. / Lucarain, L. / Fouconnier, S. / Combemale, S. / Jaquillard, L. / Schombert, B. / Chambard, J.M. / Boukaiba, R. / Hessler, G. / Bohme, A. / Bialy, L. / Hourcade, S. / Beroud, R. / De Waard, M. / Servent, D. / Partiseti, M. |
|---|
| 履歴 | | 登録 | 2018年5月2日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2019年3月6日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年4月17日 | Group: Data collection / Database references
カテゴリ: citation / citation_author / pdbx_database_proc
Item: _citation.journal_abbrev / _citation.journal_id_ISSN ..._citation.journal_abbrev / _citation.journal_id_ISSN / _citation.title / _citation_author.identifier_ORCID |
|---|
| 改定 1.2 | 2019年4月24日 | Group: Data collection / Database references / カテゴリ: citation / pdbx_database_proc
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.3 | 2019年5月8日 | Group: Data collection / カテゴリ: pdbx_nmr_software / Item: _pdbx_nmr_software.name |
|---|
| 改定 1.4 | 2023年6月14日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_nmr_spectrometer / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_nmr_spectrometer.model / _struct_conn.pdbx_dist_value |
|---|
| 改定 1.5 | 2024年10月9日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Cyriopagopus schioedtei (クモ)
Cyriopagopus schioedtei (クモ) データ登録者
データ登録者 フランス, 1件
フランス, 1件  引用
引用 ジャーナル: Br.J.Pharmacol. / 年: 2019
ジャーナル: Br.J.Pharmacol. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6gft.cif.gz
6gft.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6gft.ent.gz
pdb6gft.ent.gz PDB形式
PDB形式 6gft.json.gz
6gft.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6gft_validation.pdf.gz
6gft_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6gft_full_validation.pdf.gz
6gft_full_validation.pdf.gz 6gft_validation.xml.gz
6gft_validation.xml.gz 6gft_validation.cif.gz
6gft_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/gf/6gft
https://data.pdbj.org/pub/pdb/validation_reports/gf/6gft ftp://data.pdbj.org/pub/pdb/validation_reports/gf/6gft
ftp://data.pdbj.org/pub/pdb/validation_reports/gf/6gft リンク
リンク 集合体
集合体
 要素
要素 Cyriopagopus schioedtei (クモ) / 参照: UniProt: A0A4V8GZX0*PLUS
Cyriopagopus schioedtei (クモ) / 参照: UniProt: A0A4V8GZX0*PLUS 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj HSQC
HSQC