| 登録情報 | データベース: PDB / ID: 6fjd
|
|---|
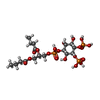
| タイトル | Human KIBRA C2 domain mutant C771A in complex with phosphatidylinositol 4,5-bisphosphate |
|---|
 要素 要素 | Protein KIBRA |
|---|
 キーワード キーワード | LIPID BINDING PROTEIN / C2 domain / Kibra / phosphoinositide-binding / membrane interaction |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of intracellular transport / regulation of hippo signaling / negative regulation of organ growth / hippo signaling / Signaling by Hippo / NOTCH3 Intracellular Domain Regulates Transcription / establishment of cell polarity / negative regulation of hippo signaling / signaling adaptor activity / kinase binding ...regulation of intracellular transport / regulation of hippo signaling / negative regulation of organ growth / hippo signaling / Signaling by Hippo / NOTCH3 Intracellular Domain Regulates Transcription / establishment of cell polarity / negative regulation of hippo signaling / signaling adaptor activity / kinase binding / ruffle membrane / cell migration / molecular adaptor activity / transcription coactivator activity / positive regulation of MAPK cascade / negative regulation of cell population proliferation / regulation of DNA-templated transcription / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II / nucleus / cytoplasm / cytosol類似検索 - 分子機能 WWC, C2 domain / : / C2 domain / C2 domain / C2 domain / C2 domain profile. / WW domain / WW/rsp5/WWP domain signature. / WW domain superfamily / WW/rsp5/WWP domain profile. ...WWC, C2 domain / : / C2 domain / C2 domain / C2 domain / C2 domain profile. / WW domain / WW/rsp5/WWP domain signature. / WW domain superfamily / WW/rsp5/WWP domain profile. / Domain with 2 conserved Trp (W) residues / WW domain / C2 domain superfamily / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.898 Å 分子置換 / 解像度: 2.898 Å |
|---|
 データ登録者 データ登録者 | Crennell, S.J. / Posner, M.G. / Bagby, S. |
|---|
| 資金援助 |  英国, 1件 英国, 1件 | 組織 | 認可番号 | 国 |
|---|
| BBSRC (Biotechnology and Biological Sciences Research Council) | BB/J008176/1 |  英国 英国 |
|
|---|
 引用 引用 |  ジャーナル: J. Biol. Chem. / 年: 2018 ジャーナル: J. Biol. Chem. / 年: 2018
タイトル: Distinctive phosphoinositide- and Ca2+-binding properties of normal and cognitive performance-linked variant forms of KIBRA C2 domain.
著者: Posner, M.G. / Upadhyay, A. / Ishima, R. / Kalli, A.C. / Harris, G. / Kremerskothen, J. / Sansom, M.S.P. / Crennell, S.J. / Bagby, S. |
|---|
| 履歴 | | 登録 | 2018年1月22日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2018年5月16日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 2.0 | 2018年6月27日 | Group: Atomic model / Data collection / Database references / カテゴリ: atom_site / citation
Item: _atom_site.occupancy / _citation.journal_volume ..._atom_site.occupancy / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title |
|---|
| 改定 2.1 | 2024年1月17日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / entity / pdbx_entity_nonpoly / pdbx_initial_refinement_model
Item: _chem_comp.name / _database_2.pdbx_DOI ..._chem_comp.name / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _entity.pdbx_description / _pdbx_entity_nonpoly.name |
|---|
| 改定 2.2 | 2024年10月23日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 2.898 Å
分子置換 / 解像度: 2.898 Å  データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: J. Biol. Chem. / 年: 2018
ジャーナル: J. Biol. Chem. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6fjd.cif.gz
6fjd.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6fjd.ent.gz
pdb6fjd.ent.gz PDB形式
PDB形式 6fjd.json.gz
6fjd.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6fjd_validation.pdf.gz
6fjd_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6fjd_full_validation.pdf.gz
6fjd_full_validation.pdf.gz 6fjd_validation.xml.gz
6fjd_validation.xml.gz 6fjd_validation.cif.gz
6fjd_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/fj/6fjd
https://data.pdbj.org/pub/pdb/validation_reports/fj/6fjd ftp://data.pdbj.org/pub/pdb/validation_reports/fj/6fjd
ftp://data.pdbj.org/pub/pdb/validation_reports/fj/6fjd リンク
リンク 集合体
集合体
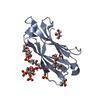

 要素
要素 Homo sapiens (ヒト) / 遺伝子: WWC1, KIAA0869 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: WWC1, KIAA0869 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54 Å
回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj