+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5zsf | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of monkey TLR7 in complex with imiquimod | |||||||||
 Components Components | Toll-like receptor 7 | |||||||||
 Keywords Keywords | IMMUNE SYSTEM / Innate immunity Toll-like receptors | |||||||||
| Function / homology |  Function and homology information Function and homology informationtoll-like receptor 7 signaling pathway / toll-like receptor 8 signaling pathway / response to cGMP / early phagosome / siRNA binding / endolysosome membrane / positive regulation of macrophage cytokine production / toll-like receptor signaling pathway / pattern recognition receptor activity / positive regulation of interferon-alpha production ...toll-like receptor 7 signaling pathway / toll-like receptor 8 signaling pathway / response to cGMP / early phagosome / siRNA binding / endolysosome membrane / positive regulation of macrophage cytokine production / toll-like receptor signaling pathway / pattern recognition receptor activity / positive regulation of interferon-alpha production / canonical NF-kappaB signal transduction / positive regulation of chemokine production / JNK cascade / positive regulation of interferon-beta production / positive regulation of interleukin-8 production / cellular response to mechanical stimulus / cellular response to virus / positive regulation of interleukin-6 production / positive regulation of type II interferon production / positive regulation of inflammatory response / double-stranded RNA binding / defense response to virus / positive regulation of canonical NF-kappaB signal transduction / signaling receptor complex / single-stranded RNA binding / inflammatory response / innate immune response / endoplasmic reticulum / positive regulation of transcription by RNA polymerase II / metal ion binding / plasma membrane Similarity search - Function | |||||||||
| Biological species |  | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.1 Å MOLECULAR REPLACEMENT / Resolution: 2.1 Å | |||||||||
 Authors Authors | Zhang, Z. / Ohto, U. / Shimizu, T. | |||||||||
 Citation Citation |  Journal: Cell Rep / Year: 2018 Journal: Cell Rep / Year: 2018Title: Structural Analyses of Toll-like Receptor 7 Reveal Detailed RNA Sequence Specificity and Recognition Mechanism of Agonistic Ligands. Authors: Zhang, Z. / Ohto, U. / Shibata, T. / Taoka, M. / Yamauchi, Y. / Sato, R. / Shukla, N.M. / David, S.A. / Isobe, T. / Miyake, K. / Shimizu, T. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5zsf.cif.gz 5zsf.cif.gz | 349 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5zsf.ent.gz pdb5zsf.ent.gz | 277.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5zsf.json.gz 5zsf.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/zs/5zsf https://data.pdbj.org/pub/pdb/validation_reports/zs/5zsf ftp://data.pdbj.org/pub/pdb/validation_reports/zs/5zsf ftp://data.pdbj.org/pub/pdb/validation_reports/zs/5zsf | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5zsaC  5zsbC  5zscC  5zsdC  5zseC  5zsgC  5zshC  5zsiC  5zsjC  5zslC  5zsmC  5zsnC  6if5C C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 2 molecules BA
| #1: Protein | Mass: 94745.594 Da / Num. of mol.: 2 / Fragment: UNP residues 27-839 / Mutation: N167Q,N389Q,N488Q,N799Q Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
|---|
-Sugars , 2 types, 14 molecules 
| #2: Polysaccharide | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose Source method: isolated from a genetically manipulated source #3: Sugar | ChemComp-NAG / |
|---|
-Non-polymers , 3 types, 870 molecules 
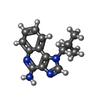



| #4: Chemical | | #5: Chemical | #6: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|---|
| Sequence details | The residues 440-445 (SEVGFC) are replaced by a thrombin-cleavage sequence (LVPRGS) for artificial ...The residues 440-445 (SEVGFC) are replaced by a thrombin-cleavage sequence (LVPRGS) for artificial cleavage of Z-loop. This engineered site in Z-loop was further cleaved during purification and the cleaved protein remained associated via interactions between LRRs and a disulfide bond. Therefore, although cleaved, the cleaved version of protein is considered as one single component (chain). |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.78 Å3/Da / Density % sol: 55.71 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, sitting drop / Details: PEG 3350, ammonium sulfate, sodium citrate pH 5.2 |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Photon Factory Photon Factory  / Beamline: AR-NE3A / Wavelength: 1 Å / Beamline: AR-NE3A / Wavelength: 1 Å |
| Detector | Type: DECTRIS PILATUS 2M-F / Detector: PIXEL / Date: Mar 12, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 2.1→50 Å / Num. obs: 123381 / % possible obs: 100 % / Redundancy: 13.5 % / Net I/σ(I): 19.9 |
| Reflection shell | Resolution: 2.1→2.14 Å |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT / Resolution: 2.1→47.161 Å / SU ML: 0.27 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 24.57 / Stereochemistry target values: ML MOLECULAR REPLACEMENT / Resolution: 2.1→47.161 Å / SU ML: 0.27 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 24.57 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.1→47.161 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller













 PDBj
PDBj







