| 登録情報 | データベース: PDB / ID: 5zc6
|
|---|
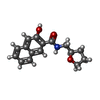
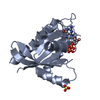
| タイトル | Solution structure of H-RasT35S mutant protein in complex with KBFM123 |
|---|
 要素 要素 | GTPase HRas |
|---|
 キーワード キーワード | ONCOPROTEIN / Inhibitor / Complex / Cancer / Small GTPases |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
phospholipase C activator activity / GTPase complex / oncogene-induced cell senescence / positive regulation of miRNA metabolic process / positive regulation of ruffle assembly / T-helper 1 type immune response / positive regulation of wound healing / defense response to protozoan / regulation of neurotransmitter receptor localization to postsynaptic specialization membrane / Signaling by RAS GAP mutants ...phospholipase C activator activity / GTPase complex / oncogene-induced cell senescence / positive regulation of miRNA metabolic process / positive regulation of ruffle assembly / T-helper 1 type immune response / positive regulation of wound healing / defense response to protozoan / regulation of neurotransmitter receptor localization to postsynaptic specialization membrane / Signaling by RAS GAP mutants / Signaling by RAS GTPase mutants / Activation of RAS in B cells / RAS signaling downstream of NF1 loss-of-function variants / SOS-mediated signalling / Activated NTRK3 signals through RAS / Activated NTRK2 signals through RAS / SHC1 events in ERBB4 signaling / positive regulation of protein targeting to membrane / Signalling to RAS / adipose tissue development / Activated NTRK2 signals through FRS2 and FRS3 / SHC-related events triggered by IGF1R / Estrogen-stimulated signaling through PRKCZ / Schwann cell development / SHC-mediated cascade:FGFR3 / MET activates RAS signaling / SHC-mediated cascade:FGFR2 / Signaling by PDGFRA transmembrane, juxtamembrane and kinase domain mutants / Signaling by PDGFRA extracellular domain mutants / PTK6 Regulates RHO GTPases, RAS GTPase and MAP kinases / SHC-mediated cascade:FGFR4 / Erythropoietin activates RAS / Signaling by FGFR4 in disease / SHC-mediated cascade:FGFR1 / FRS-mediated FGFR3 signaling / Signaling by FLT3 ITD and TKD mutants / FRS-mediated FGFR2 signaling / FRS-mediated FGFR4 signaling / protein-membrane adaptor activity / Signaling by FGFR3 in disease / p38MAPK events / FRS-mediated FGFR1 signaling / Tie2 Signaling / Signaling by FGFR2 in disease / EPHB-mediated forward signaling / GRB2 events in EGFR signaling / SHC1 events in EGFR signaling / myelination / Signaling by FLT3 fusion proteins / FLT3 Signaling / EGFR Transactivation by Gastrin / Signaling by FGFR1 in disease / NCAM signaling for neurite out-growth / CD209 (DC-SIGN) signaling / GRB2 events in ERBB2 signaling / Downstream signal transduction / Ras activation upon Ca2+ influx through NMDA receptor / intrinsic apoptotic signaling pathway / SHC1 events in ERBB2 signaling / Insulin receptor signalling cascade / Constitutive Signaling by Overexpressed ERBB2 / Signaling by phosphorylated juxtamembrane, extracellular and kinase domain KIT mutants / VEGFR2 mediated cell proliferation / animal organ morphogenesis / positive regulation of epithelial cell proliferation / small monomeric GTPase / regulation of actin cytoskeleton organization / FCERI mediated MAPK activation / positive regulation of JNK cascade / RAF activation / Signaling by ERBB2 TMD/JMD mutants / regulation of long-term neuronal synaptic plasticity / Signaling by high-kinase activity BRAF mutants / cellular response to gamma radiation / Signaling by SCF-KIT / Constitutive Signaling by EGFRvIII / MAP2K and MAPK activation / Signaling by ERBB2 ECD mutants / Signaling by ERBB2 KD Mutants / RAS processing / Regulation of RAS by GAPs / Negative regulation of MAPK pathway / positive regulation of type II interferon production / endocytosis / positive regulation of fibroblast proliferation / chemotaxis / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / GDP binding / Signaling by BRAF and RAF1 fusions / cellular senescence / Constitutive Signaling by Ligand-Responsive EGFR Cancer Variants / insulin receptor signaling pathway / DAP12 signaling / MAPK cascade / T cell receptor signaling pathway / regulation of cell population proliferation / G protein activity類似検索 - 分子機能 Small GTPase, Ras-type / Small GTPase Ras domain profile. / Rho (Ras homology) subfamily of Ras-like small GTPases / Ras subfamily of RAS small GTPases / Small GTPase / Ras family / Rab subfamily of small GTPases / Small GTP-binding protein domain / P-loop containing nucleotide triphosphate hydrolases / Rossmann fold ...Small GTPase, Ras-type / Small GTPase Ras domain profile. / Rho (Ras homology) subfamily of Ras-like small GTPases / Ras subfamily of RAS small GTPases / Small GTPase / Ras family / Rab subfamily of small GTPases / Small GTP-binding protein domain / P-loop containing nucleotide triphosphate hydrolases / Rossmann fold / P-loop containing nucleoside triphosphate hydrolase / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 PHOSPHOAMINOPHOSPHONIC ACID-GUANYLATE ESTER / Chem-KBF / GTPase HRas類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Matsumoto, S. / Hayashi, Y. / Hiraga, T. / Matsuo, K. / Kataoka, T. |
|---|
| 資金援助 |  日本, 2件 日本, 2件 | 組織 | 認可番号 | 国 |
|---|
| the Ministry of Health, Labor and Welfare and Japan Agency for Medical Research and Development | 15ak0101006h0005 |  日本 日本 | | the National Institute of Biomedical Innovation and Japan Agency for Medical Research and Development | 15nk0101401h0002 |  日本 日本 |
|
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 2018 ジャーナル: Biochemistry / 年: 2018
タイトル: Molecular Basis for Allosteric Inhibition of GTP-Bound H-Ras Protein by a Small-Molecule Compound Carrying a Naphthalene Ring
著者: Matsumoto, S. / Hiraga, T. / Hayashi, Y. / Yoshikawa, Y. / Tsuda, C. / Araki, M. / Neya, M. / Shima, F. / Kataoka, T. |
|---|
| 履歴 | | 登録 | 2018年2月15日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2018年9月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年9月26日 | Group: Data collection / Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.2 | 2024年5月1日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_spectrometer
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_spectrometer.model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 日本, 2件
日本, 2件  引用
引用 ジャーナル: Biochemistry / 年: 2018
ジャーナル: Biochemistry / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5zc6.cif.gz
5zc6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5zc6.ent.gz
pdb5zc6.ent.gz PDB形式
PDB形式 5zc6.json.gz
5zc6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5zc6_validation.pdf.gz
5zc6_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5zc6_full_validation.pdf.gz
5zc6_full_validation.pdf.gz 5zc6_validation.xml.gz
5zc6_validation.xml.gz 5zc6_validation.cif.gz
5zc6_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/zc/5zc6
https://data.pdbj.org/pub/pdb/validation_reports/zc/5zc6 ftp://data.pdbj.org/pub/pdb/validation_reports/zc/5zc6
ftp://data.pdbj.org/pub/pdb/validation_reports/zc/5zc6 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: HRAS, HRAS1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: HRAS, HRAS1 / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj



















 gel filtration
gel filtration