| 登録情報 | データベース: PDB / ID: 5z36
|
|---|
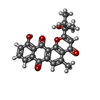
| タイトル | An anthrahydroquino-Gama-pyrone synthase Txn09 complexed with PDM |
|---|
 要素 要素 | TxnO9 |
|---|
 キーワード キーワード | BIOSYNTHETIC PROTEIN / type II polyketide heterocyclase / enzyme mechanism / natural product / Streptomyces bottropensis |
|---|
| 機能・相同性 | Activator of Hsp90 ATPase homologue 1-like / Activator of Hsp90 ATPase homolog 1-like protein / START-like domain superfamily / Chem-969 / TxnO9 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Streptomyces bottropensis (バクテリア) Streptomyces bottropensis (バクテリア) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Song, Y.J. / Cao, C.Y. |
|---|
 引用 引用 |  ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2018 ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2018
タイトル: Enzymology of Anthraquinone-gamma-Pyrone Ring Formation in Complex Aromatic Polyketide Biosynthesis.
著者: Hou, X.F. / Song, Y.J. / Zhang, M. / Lan, W.X. / Meng, S. / Wang, C.X. / Pan, H.X. / Cao, C.Y. / Tang, G.L. |
|---|
| 履歴 | | 登録 | 2018年1月5日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2018年12月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年6月14日 | Group: Data collection / Database references / Other
カテゴリ: database_2 / pdbx_database_status / pdbx_nmr_software
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_nmr_software.name |
|---|
| 改定 1.2 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Streptomyces bottropensis (バクテリア)
Streptomyces bottropensis (バクテリア) データ登録者
データ登録者 引用
引用 ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2018
ジャーナル: Angew. Chem. Int. Ed. Engl. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5z36.cif.gz
5z36.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5z36.ent.gz
pdb5z36.ent.gz PDB形式
PDB形式 5z36.json.gz
5z36.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5z36_validation.pdf.gz
5z36_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5z36_full_validation.pdf.gz
5z36_full_validation.pdf.gz 5z36_validation.xml.gz
5z36_validation.xml.gz 5z36_validation.cif.gz
5z36_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/z3/5z36
https://data.pdbj.org/pub/pdb/validation_reports/z3/5z36 ftp://data.pdbj.org/pub/pdb/validation_reports/z3/5z36
ftp://data.pdbj.org/pub/pdb/validation_reports/z3/5z36

 リンク
リンク 集合体
集合体
 要素
要素 Streptomyces bottropensis (バクテリア)
Streptomyces bottropensis (バクテリア)
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj scanning transmission electron microscopy
scanning transmission electron microscopy