Entry Database : PDB / ID : 5z25Title Trimeric Alpha-Helix-Inserted Circular Permutant of Cytochrome c555 Cytochrome c552 Keywords / Function / homology Function Domain/homology Component
Biological species Aquifex aeolicus (bacteria)Method / / / Resolution : 1.7 Å Authors Oda, A. / Nagao, S. / Yamanaka, M. / Ueda, I. / Shibata, N. / Higuchi, Y. / Hirota, S. Funding support Organization Grant number Country JSPS 26288080 JSPS 15K13744 JSPS 15H00945
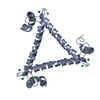
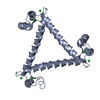
Journal : Chem Asian J / Year : 2018Title : Construction of a Triangle-Shaped Trimer and a Tetrahedron Using an alpha-Helix-Inserted Circular Permutant of Cytochrome c555.Authors : Oda, A. / Nagao, S. / Yamanaka, M. / Ueda, I. / Watanabe, H. / Uchihashi, T. / Shibata, N. / Higuchi, Y. / Hirota, S. History Deposition Dec 28, 2017 Deposition site / Processing site Revision 1.0 Mar 7, 2018 Provider / Type Revision 1.1 Mar 28, 2018 Group / Category / Item Revision 1.2 Apr 4, 2018 Group / Database references / Category / Item Revision 1.3 May 2, 2018 Group / Database references / Category Item / _citation.page_first / _citation.page_lastRevision 1.4 Nov 22, 2023 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description Category chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr1_symmetry / _pdbx_struct_conn_angle.ptnr2_symmetry / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.ptnr3_symmetry / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_symmetry Revision 1.5 Oct 30, 2024 Group / Category / pdbx_modification_feature
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information
 Aquifex aeolicus (bacteria)
Aquifex aeolicus (bacteria) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.7 Å
MOLECULAR REPLACEMENT / Resolution: 1.7 Å  Authors
Authors Japan, 3items
Japan, 3items  Citation
Citation Journal: Chem Asian J / Year: 2018
Journal: Chem Asian J / Year: 2018 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 5z25.cif.gz
5z25.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb5z25.ent.gz
pdb5z25.ent.gz PDB format
PDB format 5z25.json.gz
5z25.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads 5z25_validation.pdf.gz
5z25_validation.pdf.gz wwPDB validaton report
wwPDB validaton report 5z25_full_validation.pdf.gz
5z25_full_validation.pdf.gz 5z25_validation.xml.gz
5z25_validation.xml.gz 5z25_validation.cif.gz
5z25_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/z2/5z25
https://data.pdbj.org/pub/pdb/validation_reports/z2/5z25 ftp://data.pdbj.org/pub/pdb/validation_reports/z2/5z25
ftp://data.pdbj.org/pub/pdb/validation_reports/z2/5z25
 Links
Links Assembly
Assembly

 Components
Components
 Aquifex aeolicus (strain VF5) (bacteria)
Aquifex aeolicus (strain VF5) (bacteria)
 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  SPring-8
SPring-8  / Beamline: BL38B1 / Wavelength: 1 Å
/ Beamline: BL38B1 / Wavelength: 1 Å Processing
Processing MOLECULAR REPLACEMENT
MOLECULAR REPLACEMENT Movie
Movie Controller
Controller






 PDBj
PDBj











