+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5ytz | ||||||
|---|---|---|---|---|---|---|---|
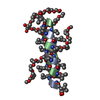
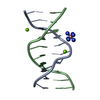
| Title | Crystal structure of echinomycin-d(ACGTCGT)2 complex | ||||||
 Components Components |
| ||||||
 Keywords Keywords | ANTIBIOTIC/DNA / Echinomycin / DNA intercalator / antibiotic / Echinomycin-DNA complex / mismatched base pairs / T-T mismatch / pyrimidine-pyrimidine base pairing / DNA mismatch repair (MMR) / non-canonical duplex structure / ANTIBIOTIC-DNA complex | ||||||
| Function / homology | Echinomycin / : / : / 2-CARBOXYQUINOXALINE / : / DNA Function and homology information Function and homology information | ||||||
| Biological species |  Streptomyces echinatus (bacteria) Streptomyces echinatus (bacteria)unidentified (others) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.55 Å MOLECULAR REPLACEMENT / Resolution: 1.55 Å | ||||||
 Authors Authors | Hou, M.H. / Wu, P.C. / Kao, Y.F. | ||||||
 Citation Citation |  Journal: Nucleic Acids Res. / Year: 2018 Journal: Nucleic Acids Res. / Year: 2018Title: Cooperative recognition of T:T mismatch by echinomycin causes structural distortions in DNA duplex Authors: Wu, P.C. / Tzeng, S.L. / Chang, C.K. / Kao, Y.F. / Waring, M.J. / Hou, M.H. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5ytz.cif.gz 5ytz.cif.gz | 42 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5ytz.ent.gz pdb5ytz.ent.gz | 28.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5ytz.json.gz 5ytz.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/yt/5ytz https://data.pdbj.org/pub/pdb/validation_reports/yt/5ytz ftp://data.pdbj.org/pub/pdb/validation_reports/yt/5ytz ftp://data.pdbj.org/pub/pdb/validation_reports/yt/5ytz | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5ytyC  3go3S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||
| Unit cell |
| ||||||||||||||||||||||||
| Components on special symmetry positions |
|
- Components
Components
-DNA chain / Protein/peptide , 2 types, 4 molecules ABDF
-Non-polymers , 4 types, 107 molecules 





| #3: Chemical | | #4: Chemical | ChemComp-QUI /   Type: Cyclic depsipeptide / Class: Antibiotic / Mass: 174.156 Da / Num. of mol.: 4 / Source method: obtained synthetically / Formula: C9H6N2O2 Type: Cyclic depsipeptide / Class: Antibiotic / Mass: 174.156 Da / Num. of mol.: 4 / Source method: obtained synthetically / Formula: C9H6N2O2Details: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ...Details: ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE. BICYCLIZATION IS ACHIEVED BY LINKING THE N- AND THE C- TERMINI, AND A THIOACETAL BOND BETWEEN RESIDUES 3 AND 7. THE TWO QUINOXALINE CHROMOPHORES ARE LINKED TO THE D-SERINE RESIDUES, RESIDUES 1 AND 5. References: Echinomycin #5: Chemical | ChemComp-K / | #6: Water | ChemComp-HOH / | |
|---|
-Details
| Compound details | THE ECHINOMYCIN IS A BICYCLIC OCTADEPSIPEPTIDE, A MEMBER OF THE QUINOXALINE CLASS OF ANTIBIOTICS. ...THE ECHINOMYCI |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.33 Å3/Da / Density % sol: 52.86 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, sitting drop Details: 20mM MES (pH 6.0), 10mM MgCl2, 2mM spermine 4HCl, 2% 2-methyl-2,4-pentanediol (MPD), 10mM MnSO4 and 10mM KBr by equilibrating against 500 ul of 30% MPD reservoir solution. |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  NSRRC NSRRC  / Beamline: TPS 05A / Wavelength: 0.92014 Å / Beamline: TPS 05A / Wavelength: 0.92014 Å |
| Detector | Type: RAYONIX MX300-HS / Detector: CCD / Date: Nov 4, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.92014 Å / Relative weight: 1 |
| Reflection | Resolution: 1.55→30 Å / Num. obs: 16665 / % possible obs: 99.9 % / Redundancy: 17.3 % / Biso Wilson estimate: 9.93 Å2 / Rmerge(I) obs: 0.037 / Rsym value: 0.037 / Net I/σ(I): 12.9 |
| Reflection shell | Resolution: 1.55→1.61 Å / Redundancy: 12.5 % / Rmerge(I) obs: 0.141 / Num. unique obs: 908 / Rsym value: 0.141 / % possible all: 99.9 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3go3 Resolution: 1.55→27.16 Å / SU ML: 0.139 / Cross valid method: FREE R-VALUE / σ(F): 1.383 / Phase error: 24.757
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 22.55 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.55→27.16 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller








 PDBj
PDBj



