| 登録情報 | データベース: PDB / ID: 5y22
|
|---|
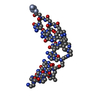
| タイトル | NMR-Based Model of the 22 Amino Acid Peptide in Polysialyltransferase Domain (PSTD) of the Polysialyltransferase ST8Sia IV |
|---|
 要素 要素 | 22AA-PSTD peptide |
|---|
 キーワード キーワード | SUGAR BINDING PROTEIN / Polysialyltransferase Domain (PSTD) Polysialyltransferase ST8Sia IV Polysialic Acid (Polysia) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Transferases; Glycosyltransferases; Sialyltransferases / alpha-N-acetylneuraminate alpha-2,8-sialyltransferase activity / sialyltransferase activity / ganglioside biosynthetic process / sialylation / Sialic acid metabolism / sialic acid binding / oligosaccharide metabolic process / N-glycan processing / NCAM1 interactions ...Transferases; Glycosyltransferases; Sialyltransferases / alpha-N-acetylneuraminate alpha-2,8-sialyltransferase activity / sialyltransferase activity / ganglioside biosynthetic process / sialylation / Sialic acid metabolism / sialic acid binding / oligosaccharide metabolic process / N-glycan processing / NCAM1 interactions / protein glycosylation / protein modification process / nervous system development / Golgi membrane / Golgi apparatus / extracellular region類似検索 - 分子機能 : / Glycosyl transferase family 29 / Sialyltransferase / GT29-like superfamiliy / Glycosyltransferase family 29 (sialyltransferase)類似検索 - ドメイン・相同性 CMP-N-acetylneuraminate-poly-alpha-2,8-sialyltransferase類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Lu, B. / Liao, S.M. / Huang, J.M. / Lu, Z.L. / Chen, D. / Liu, X.H. / Zhou, G.P. / Huang, R.B. |
|---|
| 資金援助 |  中国, 4件 中国, 4件 | 組織 | 認可番号 | 国 |
|---|
| National Science Foundation of China | 31570061 |  中国 中国 | | National Science Foundation of China | 31560251 |  中国 中国 | | National Science Foundation of Guangxi | 2015GXNSFAA139053 |  中国 中国 | | Key Research and Development Program of Guangxi | AB16380024 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: NMR-Based Model of the 22 Amino Acid Peptide in Polysialyltransferase Domain (PSTD) of the Polysialyltransferase ST8Sia IV
著者: Lu, B. / Liao, S.M. / Chen, D. / Liu, X.H. / Zhou, G.P. / Huang, R.B. |
|---|
| 履歴 | | 登録 | 2017年7月23日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2017年11月29日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年6月14日 | Group: Database references / Other / カテゴリ: database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.2 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 中国, 4件
中国, 4件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5y22.cif.gz
5y22.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5y22.ent.gz
pdb5y22.ent.gz PDB形式
PDB形式 5y22.json.gz
5y22.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5y22_validation.pdf.gz
5y22_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5y22_full_validation.pdf.gz
5y22_full_validation.pdf.gz 5y22_validation.xml.gz
5y22_validation.xml.gz 5y22_validation.cif.gz
5y22_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/y2/5y22
https://data.pdbj.org/pub/pdb/validation_reports/y2/5y22 ftp://data.pdbj.org/pub/pdb/validation_reports/y2/5y22
ftp://data.pdbj.org/pub/pdb/validation_reports/y2/5y22 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー



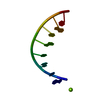
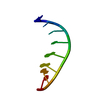


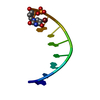
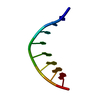
 PDBj
PDBj

 HSQC
HSQC