| 登録情報 | データベース: PDB / ID: 5ui6
|
|---|
| タイトル | Solution NMR Structure of Lasso Peptide Acinetodin |
|---|
 要素 要素 | Acinetodin |
|---|
 キーワード キーワード | ANTIMICROBIAL PROTEIN / lasso peptide RNAP inhibitor / STRUCTURE FROM CYANA 2.1 |
|---|
| 機能・相同性 | Uncharacterized protein 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Acinetobacter gyllenbergii CIP 110306T (バクテリア) Acinetobacter gyllenbergii CIP 110306T (バクテリア) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Bushin, L.B. / Metelev, M. / Severinov, K. / Seyedsayamdost, M.R. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| Searle Scholars Program | |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: ACS Chem. Biol. / 年: 2017 ジャーナル: ACS Chem. Biol. / 年: 2017
タイトル: Acinetodin and Klebsidin, RNA Polymerase Targeting Lasso Peptides Produced by Human Isolates of Acinetobacter gyllenbergii and Klebsiella pneumoniae.
著者: Metelev, M. / Arseniev, A. / Bushin, L.B. / Kuznedelov, K. / Artamonova, T.O. / Kondratenko, R. / Khodorkovskii, M. / Seyedsayamdost, M.R. / Severinov, K. |
|---|
| 履歴 | | 登録 | 2017年1月12日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年2月8日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年3月29日 | Group: Database references |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Data collection / Database references / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_nmr_ensemble / pdbx_nmr_spectrometer
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_nmr_ensemble.conformer_selection_criteria / _pdbx_nmr_spectrometer.model |
|---|
| 改定 1.3 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Acinetobacter gyllenbergii CIP 110306T (バクテリア)
Acinetobacter gyllenbergii CIP 110306T (バクテリア) データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: ACS Chem. Biol. / 年: 2017
ジャーナル: ACS Chem. Biol. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ui6.cif.gz
5ui6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ui6.ent.gz
pdb5ui6.ent.gz PDB形式
PDB形式 5ui6.json.gz
5ui6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5ui6_validation.pdf.gz
5ui6_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5ui6_full_validation.pdf.gz
5ui6_full_validation.pdf.gz 5ui6_validation.xml.gz
5ui6_validation.xml.gz 5ui6_validation.cif.gz
5ui6_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ui/5ui6
https://data.pdbj.org/pub/pdb/validation_reports/ui/5ui6 ftp://data.pdbj.org/pub/pdb/validation_reports/ui/5ui6
ftp://data.pdbj.org/pub/pdb/validation_reports/ui/5ui6
 リンク
リンク 集合体
集合体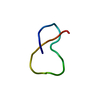
 要素
要素 Acinetobacter gyllenbergii CIP 110306T (バクテリア)
Acinetobacter gyllenbergii CIP 110306T (バクテリア) 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj HSQC
HSQC