| 登録情報 | データベース: PDB / ID: 5t7a
|
|---|
| タイトル | Crystal structure of Br derivative BhCBM56 |
|---|
 要素 要素 | BH0236 protein |
|---|
 キーワード キーワード | SUGAR BINDING PROTEIN / carbohydrate binding module |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
endo-1,3(4)-beta-glucanase activity / glucan endo-1,3-beta-D-glucosidase / glucan endo-1,3-beta-D-glucosidase activity / polysaccharide catabolic process / cell wall organization / carbohydrate binding / extracellular region類似検索 - 分子機能 Glycosyl hydrolase family 81, N-terminal / Glycosyl hydrolase family 81 N-terminal domain / Endo-1,3(4)-beta-glucanase / Glycosyl hydrolase family 81, C-terminal domain / Glycosyl hydrolase family 81 C-terminal domain / Glycosyl hydrolases family 81 (GH81) domain profile. / : / Carbohydrate binding module family 56 / CBM56 (carbohydrate binding type-56) domain profile. / Cellulose binding, type IV ...Glycosyl hydrolase family 81, N-terminal / Glycosyl hydrolase family 81 N-terminal domain / Endo-1,3(4)-beta-glucanase / Glycosyl hydrolase family 81, C-terminal domain / Glycosyl hydrolase family 81 C-terminal domain / Glycosyl hydrolases family 81 (GH81) domain profile. / : / Carbohydrate binding module family 56 / CBM56 (carbohydrate binding type-56) domain profile. / Cellulose binding, type IV / Cellulose Binding Domain Type IV / Carbohydrate binding module (family 6) / CBM6 (carbohydrate binding type-6) domain profile. / Carbohydrate binding module family 6 / Galactose-binding-like domain superfamily類似検索 - ドメイン・相同性 BROMIDE ION / Glucan endo-1,3-beta-D-glucosidase類似検索 - 構成要素 |
|---|
| 生物種 |  Bacillus halodurans (バクテリア) Bacillus halodurans (バクテリア) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 多波長異常分散 /  分子置換 / 解像度: 1.6 Å 分子置換 / 解像度: 1.6 Å |
|---|
 データ登録者 データ登録者 | Pluvinage, B. / Boraston, A.B. |
|---|
| 資金援助 |  カナダ, 1件 カナダ, 1件 | 組織 | 認可番号 | 国 |
|---|
| Natural Sciences and Engineering Research Council (NSERC, Canada) | |  カナダ カナダ |
|
|---|
 引用 引用 |  ジャーナル: J. Biol. Chem. / 年: 2017 ジャーナル: J. Biol. Chem. / 年: 2017
タイトル: Properties of a family 56 carbohydrate-binding module and its role in the recognition and hydrolysis of beta-1,3-glucan.
著者: Hettle, A. / Fillo, A. / Abe, K. / Massel, P. / Pluvinage, B. / Langelaan, D.N. / Smith, S.P. / Boraston, A.B. |
|---|
| 履歴 | | 登録 | 2016年9月2日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年8月23日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年9月27日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.2 | 2019年3月27日 | Group: Data collection / Database references / Derived calculations
カテゴリ: citation / citation_author / pdbx_struct_special_symmetry
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.name |
|---|
| 改定 1.3 | 2020年1月8日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2024年3月6日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Bacillus halodurans (バクテリア)
Bacillus halodurans (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  多波長異常分散 /
多波長異常分散 /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 カナダ, 1件
カナダ, 1件  引用
引用 ジャーナル: J. Biol. Chem. / 年: 2017
ジャーナル: J. Biol. Chem. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5t7a.cif.gz
5t7a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5t7a.ent.gz
pdb5t7a.ent.gz PDB形式
PDB形式 5t7a.json.gz
5t7a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5t7a_validation.pdf.gz
5t7a_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5t7a_full_validation.pdf.gz
5t7a_full_validation.pdf.gz 5t7a_validation.xml.gz
5t7a_validation.xml.gz 5t7a_validation.cif.gz
5t7a_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/t7/5t7a
https://data.pdbj.org/pub/pdb/validation_reports/t7/5t7a ftp://data.pdbj.org/pub/pdb/validation_reports/t7/5t7a
ftp://data.pdbj.org/pub/pdb/validation_reports/t7/5t7a リンク
リンク 集合体
集合体
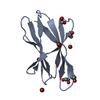

 要素
要素 Bacillus halodurans (strain ATCC BAA-125 / DSM 18197 / FERM 7344 / JCM 9153 / C-125) (バクテリア)
Bacillus halodurans (strain ATCC BAA-125 / DSM 18197 / FERM 7344 / JCM 9153 / C-125) (バクテリア)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRL
SSRL  / ビームライン: BL11-1 / 波長: 0.91966 Å
/ ビームライン: BL11-1 / 波長: 0.91966 Å 解析
解析 多波長異常分散 / 解像度: 1.6→43.73 Å / Cor.coef. Fo:Fc: 0.969 / Cor.coef. Fo:Fc free: 0.96 / SU B: 1.502 / SU ML: 0.053 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.085 / ESU R Free: 0.084
多波長異常分散 / 解像度: 1.6→43.73 Å / Cor.coef. Fo:Fc: 0.969 / Cor.coef. Fo:Fc free: 0.96 / SU B: 1.502 / SU ML: 0.053 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.085 / ESU R Free: 0.084  ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj






