| 登録情報 | データベース: PDB / ID: 5ow8
|
|---|
| タイトル | Indole-2 carboxamides as selective secreted phospholipase A2 type X (sPLA2-X) inhibitors |
|---|
 要素 要素 | Group 10 secretory phospholipase A2 |
|---|
 キーワード キーワード | LIPID BINDING PROTEIN / Inhibitor / secreted / phospholipase |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
nuclear receptor-mediated signaling pathway / phosphatidylserine metabolic process / phosphatidylethanolamine metabolic process / lysophospholipid transport / phosphatidic acid metabolic process / platelet activating factor catabolic process / positive regulation of acrosome reaction / Acyl chain remodelling of PG / Acyl chain remodelling of PC / Acyl chain remodelling of PI ...nuclear receptor-mediated signaling pathway / phosphatidylserine metabolic process / phosphatidylethanolamine metabolic process / lysophospholipid transport / phosphatidic acid metabolic process / platelet activating factor catabolic process / positive regulation of acrosome reaction / Acyl chain remodelling of PG / Acyl chain remodelling of PC / Acyl chain remodelling of PI / Acyl chain remodelling of PS / Acyl chain remodelling of PE / phospholipase activity / 1-alkyl-2-acetylglycerophosphocholine esterase activity / Synthesis of PA / production of molecular mediator involved in inflammatory response / negative regulation of cholesterol efflux / intestinal stem cell homeostasis / arachidonate metabolic process / phosphatidylglycerol metabolic process / phospholipase A2 activity / phosphatidylcholine metabolic process / positive regulation of prostaglandin secretion / positive regulation of arachidonate secretion / macrophage activation / positive regulation of lipid storage / phospholipase A2 / low-density lipoprotein particle remodeling / positive regulation of macrophage derived foam cell differentiation / phosphatidylcholine catabolic process / calcium-dependent phospholipase A2 activity / fertilization / prostaglandin biosynthetic process / signal transduction involved in regulation of gene expression / arachidonate secretion / hair follicle morphogenesis / regulation of macrophage activation / erythrocyte maturation / positive regulation of protein metabolic process / negative regulation of cytokine production involved in inflammatory response / acrosomal vesicle / axon guidance / cholesterol homeostasis / cellular response to leukemia inhibitory factor / phospholipid binding / negative regulation of inflammatory response / defense response to virus / lysosome / calcium ion binding / negative regulation of transcription by RNA polymerase II / extracellular space / extracellular region類似検索 - 分子機能 Phospholipase A2, aspartic acid active site / Phospholipase A2 aspartic acid active site. / Phospholipase A2 / Phospholipase A2 / Phospholipase A2, histidine active site / Phospholipase A2 histidine active site. / Phospholipase A2 domain / Phospholipase A2 / Phospholipase A2 / Phospholipase A2 domain ...Phospholipase A2, aspartic acid active site / Phospholipase A2 aspartic acid active site. / Phospholipase A2 / Phospholipase A2 / Phospholipase A2, histidine active site / Phospholipase A2 histidine active site. / Phospholipase A2 domain / Phospholipase A2 / Phospholipase A2 / Phospholipase A2 domain / Phospholipase A2 domain superfamily / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 1-[3-(trifluoromethyl)phenyl]indole-2-carboxamide / DI(HYDROXYETHYL)ETHER / Group 10 secretory phospholipase A2類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Sandmark, J.S. / Roth, R.G. / Knerr, L. / Bodin, C. / Pettersen, D. |
|---|
 引用 引用 |  ジャーナル: Acs Med.Chem.Lett. / 年: 2018 ジャーナル: Acs Med.Chem.Lett. / 年: 2018
タイトル: Discovery of a Series of Indole-2 Carboxamides as Selective Secreted Phospholipase A2Type X (sPLA2-X) Inhibitors.
著者: Knerr, L. / Giordanetto, F. / Nordberg, P. / Pettersen, D. / Selmi, N. / Beisel, H.G. / de la Motte, H. / Olsson, T. / Perkins, T.D.J. / Herslof, M. / Mansson, A. / Dahlstrom, M. / Starke, I. ...著者: Knerr, L. / Giordanetto, F. / Nordberg, P. / Pettersen, D. / Selmi, N. / Beisel, H.G. / de la Motte, H. / Olsson, T. / Perkins, T.D.J. / Herslof, M. / Mansson, A. / Dahlstrom, M. / Starke, I. / Broddefalk, J. / Saarinen, G. / Klingegard, F. / Hurt-Camejo, E. / Rosengren, B. / Brengdahl, J. / Jansen, F. / Rohman, M. / Sandmark, J. / Hallberg, K. / Akerud, T. / Roth, R.G. / Ahlqvist, M. |
|---|
| 履歴 | | 登録 | 2017年8月31日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2018年8月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2021年5月5日 | Group: Database references / Derived calculations
カテゴリ: citation / pdbx_struct_assembly ...citation / pdbx_struct_assembly / pdbx_struct_assembly_gen / pdbx_struct_assembly_prop / pdbx_struct_conn_angle / pdbx_struct_oper_list / struct_conn
Item: _citation.journal_abbrev / _pdbx_struct_assembly_gen.assembly_id ..._citation.journal_abbrev / _pdbx_struct_assembly_gen.assembly_id / _pdbx_struct_assembly_gen.oper_expression / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id |
|---|
| 改定 1.2 | 2024年10月16日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Acs Med.Chem.Lett. / 年: 2018
ジャーナル: Acs Med.Chem.Lett. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ow8.cif.gz
5ow8.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ow8.ent.gz
pdb5ow8.ent.gz PDB形式
PDB形式 5ow8.json.gz
5ow8.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5ow8_validation.pdf.gz
5ow8_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5ow8_full_validation.pdf.gz
5ow8_full_validation.pdf.gz 5ow8_validation.xml.gz
5ow8_validation.xml.gz 5ow8_validation.cif.gz
5ow8_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ow/5ow8
https://data.pdbj.org/pub/pdb/validation_reports/ow/5ow8 ftp://data.pdbj.org/pub/pdb/validation_reports/ow/5ow8
ftp://data.pdbj.org/pub/pdb/validation_reports/ow/5ow8 リンク
リンク 集合体
集合体
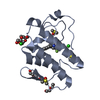

 要素
要素 Homo sapiens (ヒト) / 遺伝子: PLA2G10 / プラスミド: pET24 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PLA2G10 / プラスミド: pET24 / 発現宿主: 


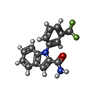








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E+ DW / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU FR-E+ DW / 波長: 1.5418 Å 解析
解析 分子置換 / 解像度: 1.9→44 Å / Cor.coef. Fo:Fc: 0.931 / Cor.coef. Fo:Fc free: 0.898 / SU B: 4.589 / SU ML: 0.13 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.216 / ESU R Free: 0.179
分子置換 / 解像度: 1.9→44 Å / Cor.coef. Fo:Fc: 0.931 / Cor.coef. Fo:Fc free: 0.898 / SU B: 4.589 / SU ML: 0.13 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.216 / ESU R Free: 0.179  ムービー
ムービー コントローラー
コントローラー



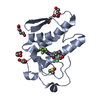










 PDBj
PDBj






