[English] 日本語
 Yorodumi
Yorodumi- PDB-5orr: Crystal structure of Aurora-A kinase in complex with an allosteri... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5orr | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of Aurora-A kinase in complex with an allosterically binding fragment | |||||||||
 Components Components | Aurora kinase A | |||||||||
 Keywords Keywords | TRANSFERASE / kinase / allosteric inhibitor / fragment | |||||||||
| Function / homology |  Function and homology information Function and homology informationInteraction between PHLDA1 and AURKA / regulation of centrosome cycle / axon hillock / spindle assembly involved in female meiosis I / cilium disassembly / spindle pole centrosome / chromosome passenger complex / histone H3S10 kinase activity / positive regulation of oocyte maturation / mitotic centrosome separation ...Interaction between PHLDA1 and AURKA / regulation of centrosome cycle / axon hillock / spindle assembly involved in female meiosis I / cilium disassembly / spindle pole centrosome / chromosome passenger complex / histone H3S10 kinase activity / positive regulation of oocyte maturation / mitotic centrosome separation / pronucleus / germinal vesicle / protein localization to centrosome / meiotic spindle / anterior/posterior axis specification / neuron projection extension / spindle organization / centrosome localization / positive regulation of mitochondrial fission / mitotic spindle pole / spindle midzone / SUMOylation of DNA replication proteins / negative regulation of protein binding / regulation of G2/M transition of mitotic cell cycle / positive regulation of mitotic nuclear division / protein serine/threonine/tyrosine kinase activity / centriole / liver regeneration / positive regulation of mitotic cell cycle / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / molecular function activator activity / AURKA Activation by TPX2 / regulation of signal transduction by p53 class mediator / mitotic spindle organization / regulation of cytokinesis / peptidyl-serine phosphorylation / regulation of protein stability / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / kinetochore / response to wounding / G2/M transition of mitotic cell cycle / spindle / spindle pole / mitotic spindle / Regulation of PLK1 Activity at G2/M Transition / mitotic cell cycle / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / protein autophosphorylation / microtubule cytoskeleton / midbody / basolateral plasma membrane / Regulation of TP53 Activity through Phosphorylation / microtubule / proteasome-mediated ubiquitin-dependent protein catabolic process / protein phosphorylation / protein kinase activity / non-specific serine/threonine protein kinase / postsynaptic density / ciliary basal body / protein heterodimerization activity / negative regulation of gene expression / cell division / protein serine kinase activity / protein serine/threonine kinase activity / apoptotic process / ubiquitin protein ligase binding / centrosome / protein kinase binding / negative regulation of apoptotic process / perinuclear region of cytoplasm / glutamatergic synapse / nucleoplasm / ATP binding / nucleus / cytosol Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / Resolution: 2.09 Å SYNCHROTRON / Resolution: 2.09 Å | |||||||||
 Authors Authors | McIntyre, P.J. / Collins, P.M. / von Delft, F. / Bayliss, R. | |||||||||
| Funding support |  United Kingdom, 2items United Kingdom, 2items
| |||||||||
 Citation Citation |  Journal: ACS Chem. Biol. / Year: 2017 Journal: ACS Chem. Biol. / Year: 2017Title: Characterization of Three Druggable Hot-Spots in the Aurora-A/TPX2 Interaction Using Biochemical, Biophysical, and Fragment-Based Approaches. Authors: McIntyre, P.J. / Collins, P.M. / Vrzal, L. / Birchall, K. / Arnold, L.H. / Mpamhanga, C. / Coombs, P.J. / Burgess, S.G. / Richards, M.W. / Winter, A. / Veverka, V. / Delft, F.V. / Merritt, A. / Bayliss, R. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5orr.cif.gz 5orr.cif.gz | 129.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5orr.ent.gz pdb5orr.ent.gz | 98.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5orr.json.gz 5orr.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/or/5orr https://data.pdbj.org/pub/pdb/validation_reports/or/5orr ftp://data.pdbj.org/pub/pdb/validation_reports/or/5orr ftp://data.pdbj.org/pub/pdb/validation_reports/or/5orr | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5orlC  5ornC  5oroC  5orpC  5orsC  5ortC  5orvC  5orwC  5orxC  5oryC  5orzC  5os0C  5os1C  5os2C  5os3C  5os4C  5os5C  5os6C  5osdC  5oseC  5osfC C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 1 molecules A
| #1: Protein | Mass: 30798.236 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) Homo sapiens (human)Gene: AURKA, AIK, AIRK1, ARK1, AURA, AYK1, BTAK, IAK1, STK15, STK6 Production host:  References: UniProt: O14965, non-specific serine/threonine protein kinase |
|---|
-Non-polymers , 5 types, 105 molecules 

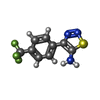






| #2: Chemical | ChemComp-ADP / | ||||||
|---|---|---|---|---|---|---|---|
| #3: Chemical | | #4: Chemical | ChemComp-A5Q / | #5: Chemical | #6: Water | ChemComp-HOH / | |
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.79 Å3/Da / Density % sol: 55.92 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, hanging drop / pH: 8.5 Details: 0.1 M Tris, pH 8.5; 0.5 M NaCl; 0.2 M MgCl2; 32.5 % v/v PEG 3350 |
-Data collection
| Diffraction | Mean temperature: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Diamond Diamond  / Beamline: I04-1 / Wavelength: 0.9282 Å / Beamline: I04-1 / Wavelength: 0.9282 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: DECTRIS PILATUS 6M-F / Detector: PIXEL / Date: Feb 15, 2016 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9282 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 2.09→65.978 Å / Num. obs: 39499 / % possible obs: 100 % / Observed criterion σ(I): -3 / Redundancy: 10.027 % / Biso Wilson estimate: 52.611 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.115 / Rrim(I) all: 0.122 / Χ2: 1.017 / Net I/σ(I): 13.43 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Diffraction-ID: 1
|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Resolution: 2.09→65.978 Å / SU ML: 0.3 / Cross valid method: FREE R-VALUE / σ(F): 1.35 / Phase error: 25.06
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 155.44 Å2 / Biso mean: 52.7137 Å2 / Biso min: 29.54 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 2.09→65.978 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller






















 PDBj
PDBj













