[English] 日本語
 Yorodumi
Yorodumi- PDB-5o43: 17beta-hydroxysteroid dehydrogenase 14 variant T205 in complex wi... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5o43 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | 17beta-hydroxysteroid dehydrogenase 14 variant T205 in complex with a non-steroidal 2,6-pyridinketone inhibitor. | |||||||||
 Components Components | 17-beta-hydroxysteroid dehydrogenase 14 | |||||||||
 Keywords Keywords | OXIDOREDUCTASE / non-steroidal inhibitor / 17beta-HSD14 / 2 / 6-pyridinketone inhibitors | |||||||||
| Function / homology |  Function and homology information Function and homology informationD-threo-aldose 1-dehydrogenase / D-threo-aldose 1-dehydrogenase activity / Estrogen biosynthesis / L-fucose catabolic process / estradiol 17-beta-dehydrogenase [NAD(P)+] activity / steroid catabolic process / identical protein binding / cytosol Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.5 Å MOLECULAR REPLACEMENT / Resolution: 1.5 Å | |||||||||
 Authors Authors | Bertoletti, N. / Heine, A. / Braun, F. / Klebe, G. / Marchais-Oberwinkler, S. | |||||||||
| Funding support |  Germany, 2items Germany, 2items
| |||||||||
 Citation Citation |  Journal: Eur J Med Chem / Year: 2018 Journal: Eur J Med Chem / Year: 2018Title: Structure-based design and profiling of novel 17 beta-HSD14 inhibitors. Authors: Braun, F. / Bertoletti, N. / Moller, G. / Adamski, J. / Frotscher, M. / Guragossian, N. / Madeira Girio, P.A. / Le Borgne, M. / Ettouati, L. / Falson, P. / Muller, S. / Vollmer, G. / Heine, ...Authors: Braun, F. / Bertoletti, N. / Moller, G. / Adamski, J. / Frotscher, M. / Guragossian, N. / Madeira Girio, P.A. / Le Borgne, M. / Ettouati, L. / Falson, P. / Muller, S. / Vollmer, G. / Heine, A. / Klebe, G. / Marchais-Oberwinkler, S. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5o43.cif.gz 5o43.cif.gz | 153.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5o43.ent.gz pdb5o43.ent.gz | 120.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5o43.json.gz 5o43.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/o4/5o43 https://data.pdbj.org/pub/pdb/validation_reports/o4/5o43 ftp://data.pdbj.org/pub/pdb/validation_reports/o4/5o43 ftp://data.pdbj.org/pub/pdb/validation_reports/o4/5o43 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5o42C  5o6oC  5o6xC  5o6zC  5o72C  5o7cC  5en4S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| Unit cell |
| ||||||||||||
| Components on special symmetry positions |
|
- Components
Components
-Protein / Sugars , 2 types, 2 molecules A

| #1: Protein | Mass: 28682.740 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: HSD17B14, DHRS10, SDR3, SDR47C1, UNQ502/PRO474 / Production host: Homo sapiens (human) / Gene: HSD17B14, DHRS10, SDR3, SDR47C1, UNQ502/PRO474 / Production host:  References: UniProt: Q9BPX1, Oxidoreductases; Acting on the CH-OH group of donors; With NAD+ or NADP+ as acceptor |
|---|---|
| #4: Sugar | ChemComp-BGC / |
-Non-polymers , 4 types, 223 molecules 

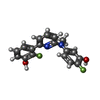




| #2: Chemical | ChemComp-NA / |
|---|---|
| #3: Chemical | ChemComp-NAD / |
| #5: Chemical | ChemComp-9JQ / |
| #6: Water | ChemComp-HOH / |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.43 Å3/Da / Density % sol: 49.42 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion, sitting drop / Details: PEG 6000 20% Hepes 0.1M DMSO 5% / PH range: 7-8 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  BESSY BESSY  / Beamline: 14.1 / Wavelength: 0.9184 Å / Beamline: 14.1 / Wavelength: 0.9184 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Dec 10, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9184 Å / Relative weight: 1 |
| Reflection | Resolution: 1.5→50 Å / Num. obs: 45487 / % possible obs: 99.9 % / Redundancy: 8.53 % / Rsym value: 0.09 / Net I/σ(I): 13.67 |
| Reflection shell | Resolution: 1.5→1.59 Å / Redundancy: 8.8 % / Mean I/σ(I) obs: 3.3 / Num. unique all: 7233 / CC1/2: 0.938 / Rsym value: 0.504 / % possible all: 100 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5EN4 Resolution: 1.5→50 Å / SU ML: 0.1 / Cross valid method: FREE R-VALUE / σ(F): 1.36 / Phase error: 16.49
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.5→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj









