| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| PHENIX | (dev_2219: ???)| 精密化 | | xia2 | | データ削減 | | xia2 | | データスケーリング | | PHASER | | 位相決定 | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: 5NWQ
解像度: 2.94→32.938 Å / SU ML: 0.49 / 交差検証法: THROUGHOUT / σ(F): 1.33 / 位相誤差: 30.5
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.2169 | 789 | 4.88 % |
|---|
| Rwork | 0.1912 | - | - |
|---|
| obs | 0.1924 | 16177 | 98.56 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å |
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 2.94→32.938 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 0 | 882 | 4 | 0 | 886 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.009 | 993 | | X-RAY DIFFRACTION | f_angle_d| 1.721 | 1550 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 18.285 | 469 | | X-RAY DIFFRACTION | f_chiral_restr| 0.07 | 202 | | X-RAY DIFFRACTION | f_plane_restr| 0.01 | 42 | | | | | |
|
|---|
| LS精密化 シェル | | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Refine-ID | % reflection obs (%) |
|---|
| 2.94-3.1241 | 0.4682 | 149 | 0.4107 | 2488 | X-RAY DIFFRACTION | 97 | | 3.1241-3.3651 | 0.2567 | 119 | 0.2268 | 2571 | X-RAY DIFFRACTION | 98 | | 3.3651-3.7033 | 0.2159 | 152 | 0.2101 | 2538 | X-RAY DIFFRACTION | 99 | | 3.7033-4.2383 | 0.2014 | 148 | 0.2007 | 2551 | X-RAY DIFFRACTION | 99 | | 4.2383-5.3361 | 0.2228 | 90 | 0.1647 | 2616 | X-RAY DIFFRACTION | 99 | | 5.3361-32.94 | 0.1721 | 131 | 0.1574 | 2624 | X-RAY DIFFRACTION | 100 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 1.0117 | -1.01 | -0.734 | 7.8488 | -0.4022 | 0.6486 | 0.1168 | -0.9113 | 0.3122 | 0.2831 | -0.0993 | 1.1979 | -0.3561 | 0.2593 | -0.0137 | 1.0534 | -0.448 | -0.1971 | 1.0217 | 0.1308 | 0.8361 | -15.9453 | -11.5367 | 13.3855 | | 2 | 2.0028 | 1.706 | 0.0464 | 3.2444 | -0.3404 | 1.4766 | -0.098 | -0.4993 | -0.1275 | -0.173 | -0.0857 | -0.2104 | -0.3518 | 0.4247 | 0.2232 | 1.1593 | -0.4348 | -0.0906 | 0.9236 | 0.0017 | 0.9338 | -19.9275 | -19.8592 | 15.2363 | | 3 | 4.9682 | -0.3633 | -1.591 | 7.3736 | -1.1503 | 4.3941 | 1.1341 | -1.0148 | -0.0633 | 0.3373 | -0.7924 | -0.3976 | -0.8561 | 0.9552 | -0.2907 | 1.4416 | -0.4805 | -0.1727 | 0.9645 | 0.0029 | 1.1012 | -9.8593 | 2.0031 | 11.1299 | | 4 | 6.7589 | 0.7068 | -3.9319 | 1.3726 | 0.0683 | 8.6557 | -0.2059 | 1.0512 | 0.9416 | 0.8404 | 0.0581 | -0.7922 | -0.6338 | 0.5787 | -0.1605 | 0.9085 | -0.5316 | -0.2416 | 1.0174 | -0.0103 | 0.5251 | -11.9178 | -1.6722 | 4.035 | | 5 | 3.1091 | 1.0782 | -1.2553 | 1.3616 | -0.9433 | 4.5677 | 0.2311 | -0.4317 | -0.7938 | 0.8089 | -1.0965 | -1.2338 | 0.7385 | -0.2333 | 0.6469 | 0.9279 | -0.2521 | -0.155 | 1.0551 | 0.2582 | 0.8867 | -5.8553 | -15.6292 | 11.8538 | | 6 | 6.6749 | -0.2094 | 0.1144 | 6.5967 | -0.6593 | 5.4475 | 0.9484 | -0.5779 | -0.3942 | 0.2163 | -0.4467 | 1.8989 | 0.1375 | 0.501 | -0.2436 | 1.3779 | -0.4424 | 0.1745 | 0.6761 | 0.025 | 0.8887 | -24.7248 | -21.4474 | 20.0191 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details |
|---|
| 1 | X-RAY DIFFRACTION | 1 | chain 'A' and (resid 1 through 10 )| 2 | X-RAY DIFFRACTION | 2 | chain 'A' and (resid 11 through 20 )| 3 | X-RAY DIFFRACTION | 3 | chain 'A' and (resid 21 through 25 )| 4 | X-RAY DIFFRACTION | 4 | chain 'A' and (resid 26 through 30 )| 5 | X-RAY DIFFRACTION | 5 | chain 'A' and (resid 31 through 35 )| 6 | X-RAY DIFFRACTION | 6 | chain 'A' and (resid 36 through 41 ) | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
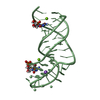
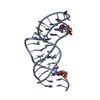
 Thermobifida fusca (バクテリア)
Thermobifida fusca (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.94 Å
分子置換 / 解像度: 2.94 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell Chem Biol / 年: 2017
ジャーナル: Cell Chem Biol / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5nz6.cif.gz
5nz6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5nz6.ent.gz
pdb5nz6.ent.gz PDB形式
PDB形式 5nz6.json.gz
5nz6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5nz6_validation.pdf.gz
5nz6_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5nz6_full_validation.pdf.gz
5nz6_full_validation.pdf.gz 5nz6_validation.xml.gz
5nz6_validation.xml.gz 5nz6_validation.cif.gz
5nz6_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/nz/5nz6
https://data.pdbj.org/pub/pdb/validation_reports/nz/5nz6 ftp://data.pdbj.org/pub/pdb/validation_reports/nz/5nz6
ftp://data.pdbj.org/pub/pdb/validation_reports/nz/5nz6 リンク
リンク 集合体
集合体
 要素
要素
 Thermobifida fusca (バクテリア)
Thermobifida fusca (バクテリア) X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.92019 Å
/ ビームライン: I03 / 波長: 0.92019 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj