| 登録情報 | データベース: PDB / ID: 5mnw
|
|---|
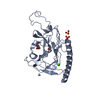
| タイトル | Solution structure of the cinaciguat bound human beta1 H-NOX. |
|---|
 要素 要素 | Guanylate cyclase soluble subunit beta-1 |
|---|
 キーワード キーワード | LYASE / soluble guanylate cyclase / H-NOX / cinaciguat |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cytidylate cyclase activity / guanylate cyclase complex, soluble / guanylate cyclase / cGMP biosynthetic process / guanylate cyclase activity / presynaptic active zone cytoplasmic component / response to oxygen levels / nitric oxide binding / Nitric oxide stimulates guanylate cyclase / adenylate cyclase activity ...cytidylate cyclase activity / guanylate cyclase complex, soluble / guanylate cyclase / cGMP biosynthetic process / guanylate cyclase activity / presynaptic active zone cytoplasmic component / response to oxygen levels / nitric oxide binding / Nitric oxide stimulates guanylate cyclase / adenylate cyclase activity / blood circulation / cGMP-mediated signaling / nitric oxide mediated signal transduction / Smooth Muscle Contraction / nitric oxide-cGMP-mediated signaling / cellular response to nitric oxide / Hsp90 protein binding / signaling receptor activity / heme binding / GTP binding / protein-containing complex binding / glutamatergic synapse / metal ion binding / cytosol類似検索 - 分子機能 Haem NO binding associated / Haem NO binding associated domain superfamily / Heme NO binding associated / Heme NO-binding / H-NOX domain superfamily / Haem-NO-binding / Adenylyl cyclase class-4/guanylyl cyclase, conserved site / Guanylate cyclase signature. / NO signalling/Golgi transport ligand-binding domain superfamily / Adenylyl- / guanylyl cyclase, catalytic domain ...Haem NO binding associated / Haem NO binding associated domain superfamily / Heme NO binding associated / Heme NO-binding / H-NOX domain superfamily / Haem-NO-binding / Adenylyl cyclase class-4/guanylyl cyclase, conserved site / Guanylate cyclase signature. / NO signalling/Golgi transport ligand-binding domain superfamily / Adenylyl- / guanylyl cyclase, catalytic domain / Adenylate and Guanylate cyclase catalytic domain / Adenylyl cyclase class-3/4/guanylyl cyclase / Guanylate cyclase domain profile. / Nucleotide cyclase類似検索 - ドメイン・相同性 Chem-Z90 / Guanylate cyclase soluble subunit beta-1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Matzapetakis, M. / Saraiva, I.H. |
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Solution structure of the cinaciguat bound human beta1 H-NOX.
著者: Matzapetakis, M. / Saraiva, I.H. |
|---|
| 履歴 | | 登録 | 2016年12月13日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2018年6月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年5月8日 | Group: Data collection / カテゴリ: pdbx_nmr_software / Item: _pdbx_nmr_software.name |
|---|
| 改定 1.2 | 2024年6月19日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_spectrometer
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_spectrometer.model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5mnw.cif.gz
5mnw.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5mnw.ent.gz
pdb5mnw.ent.gz PDB形式
PDB形式 5mnw.json.gz
5mnw.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5mnw_validation.pdf.gz
5mnw_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5mnw_full_validation.pdf.gz
5mnw_full_validation.pdf.gz 5mnw_validation.xml.gz
5mnw_validation.xml.gz 5mnw_validation.cif.gz
5mnw_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/mn/5mnw
https://data.pdbj.org/pub/pdb/validation_reports/mn/5mnw ftp://data.pdbj.org/pub/pdb/validation_reports/mn/5mnw
ftp://data.pdbj.org/pub/pdb/validation_reports/mn/5mnw リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: GUCY1B3, GUC1B3, GUCSB3, GUCY1B1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: GUCY1B3, GUC1B3, GUCSB3, GUCY1B1 / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj







 HSQC
HSQC