| 登録情報 | データベース: PDB / ID: 5lqv
|
|---|
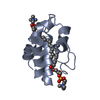
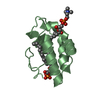
| タイトル | Spatial structure of the lentil lipid transfer protein in complex with anionic lysolipid LPPG |
|---|
 要素 要素 | Non-specific lipid-transfer protein 2 |
|---|
 キーワード キーワード | LIPID TRANSPORT / plant defense peptide lipid transfer protein lens culinaris complex with lipid / STRUCTURE FROM CYANA 3.0 / lipid transfer |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Plant lipid transfer proteins signature. / Plant non-specific lipid-transfer protein/Par allergen / Plant lipid-transfer and hydrophobic proteins / Hydrophobic Seed Protein / Protease inhibitor/seed storage/LTP family / Plant lipid transfer protein / seed storage protein / trypsin-alpha amylase inhibitor domain family / Bifunctional inhibitor/plant lipid transfer protein/seed storage helical domain / Bifunctional inhibitor/plant lipid transfer protein/seed storage helical domain superfamily / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 Chem-PGM / Non-specific lipid-transfer protein 2類似検索 - 構成要素 |
|---|
| 生物種 |  Lens culinaris (マメ科) Lens culinaris (マメ科) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Mineev, K.S. / Shenkarev, Z.O. / Arseniev, A.S. / Melnikova, D.N. / Finkina, E.I. / Ovchinnikova, T.V. |
|---|
| 資金援助 |  ロシア, 1件 ロシア, 1件 | 組織 | 認可番号 | 国 |
|---|
| Russian Science Foundation | 14-50-00131 |  ロシア ロシア |
|
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 2017 ジャーナル: Biochemistry / 年: 2017
タイトル: Ligand Binding Properties of the Lentil Lipid Transfer Protein: Molecular Insight into the Possible Mechanism of Lipid Uptake.
著者: Shenkarev, Z.O. / Melnikova, D.N. / Finkina, E.I. / Sukhanov, S.V. / Boldyrev, I.A. / Gizatullina, A.K. / Mineev, K.S. / Arseniev, A.S. / Ovchinnikova, T.V. |
|---|
| 履歴 | | 登録 | 2016年8月17日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年6月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年5月8日 | Group: Data collection / カテゴリ: pdbx_nmr_software / Item: _pdbx_nmr_software.name |
|---|
| 改定 1.2 | 2024年11月13日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_nmr_spectrometer
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_spectrometer.model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Lens culinaris (マメ科)
Lens culinaris (マメ科) データ登録者
データ登録者 ロシア, 1件
ロシア, 1件  引用
引用 ジャーナル: Biochemistry / 年: 2017
ジャーナル: Biochemistry / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5lqv.cif.gz
5lqv.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5lqv.ent.gz
pdb5lqv.ent.gz PDB形式
PDB形式 5lqv.json.gz
5lqv.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5lqv_validation.pdf.gz
5lqv_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5lqv_full_validation.pdf.gz
5lqv_full_validation.pdf.gz 5lqv_validation.xml.gz
5lqv_validation.xml.gz 5lqv_validation.cif.gz
5lqv_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/lq/5lqv
https://data.pdbj.org/pub/pdb/validation_reports/lq/5lqv ftp://data.pdbj.org/pub/pdb/validation_reports/lq/5lqv
ftp://data.pdbj.org/pub/pdb/validation_reports/lq/5lqv リンク
リンク 集合体
集合体
 要素
要素 Lens culinaris (マメ科) / 発現宿主:
Lens culinaris (マメ科) / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


 HSQC
HSQC