| 登録情報 | データベース: PDB / ID: 5lo8
|
|---|
| タイトル | The C2B domain of Rabphilin 3A in complex with PI(4,5)P2 |
|---|
 要素 要素 | Rabphilin-3A |
|---|
 キーワード キーワード | PROTEIN TRANSPORT / vesicle fusion / PIP2 / C2 domain |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
selenium binding / spontaneous neurotransmitter secretion / calcium-dependent activation of synaptic vesicle fusion / cholinergic synapse / positive regulation of calcium ion-dependent exocytosis / extrinsic component of synaptic vesicle membrane / inositol 1,4,5 trisphosphate binding / calcium-dependent phospholipid binding / dendritic spine organization / regulation of NMDA receptor activity ...selenium binding / spontaneous neurotransmitter secretion / calcium-dependent activation of synaptic vesicle fusion / cholinergic synapse / positive regulation of calcium ion-dependent exocytosis / extrinsic component of synaptic vesicle membrane / inositol 1,4,5 trisphosphate binding / calcium-dependent phospholipid binding / dendritic spine organization / regulation of NMDA receptor activity / extrinsic component of membrane / synaptic vesicle priming / phosphate ion binding / phosphatidylinositol-4,5-bisphosphate binding / secretory granule / intracellular protein transport / neuromuscular junction / phospholipid binding / small GTPase binding / synaptic vesicle / synaptic vesicle membrane / dendritic spine / postsynaptic membrane / neuron projection / synapse / calcium ion binding / protein-containing complex binding / protein-containing complex / zinc ion binding類似検索 - 分子機能 Rabphilin-3A / Rabphilin/DOC2/Noc2 / : / FYVE-type zinc finger / FYVE-type zinc finger / Rab-binding domain / Rab-binding domain profile. / Zinc finger, FYVE-related / Zinc finger FYVE/FYVE-related type profile. / Synaptotagmin ...Rabphilin-3A / Rabphilin/DOC2/Noc2 / : / FYVE-type zinc finger / FYVE-type zinc finger / Rab-binding domain / Rab-binding domain profile. / Zinc finger, FYVE-related / Zinc finger FYVE/FYVE-related type profile. / Synaptotagmin / C2 domain / Protein kinase C conserved region 2 (CalB) / C2 domain / C2 domain / C2 domain profile. / C2 domain superfamily / Zinc finger, FYVE/PHD-type / Zinc finger, RING/FYVE/PHD-type / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å |
|---|
 データ登録者 データ登録者 | Ferrer-Orta, C. / Verdaguer, N. |
|---|
 引用 引用 |  ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017
タイトル: Structural characterization of the Rabphilin-3A-SNAP25 interaction.
著者: Ferrer-Orta, C. / Perez-Sanchez, M.D. / Coronado-Parra, T. / Silva, C. / Lopez-Martinez, D. / Baltanas-Copado, J. / Gomez-Fernandez, J.C. / Corbalan-Garcia, S. / Verdaguer, N. |
|---|
| 履歴 | | 登録 | 2016年8月8日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年6月21日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年12月6日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.name |
|---|
| 改定 1.2 | 2024年1月10日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn / struct_conn_type
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_symmetry / _struct_conn_type.id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.5 Å
分子置換 / 解像度: 2.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017
ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5lo8.cif.gz
5lo8.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5lo8.ent.gz
pdb5lo8.ent.gz PDB形式
PDB形式 5lo8.json.gz
5lo8.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5lo8_validation.pdf.gz
5lo8_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5lo8_full_validation.pdf.gz
5lo8_full_validation.pdf.gz 5lo8_validation.xml.gz
5lo8_validation.xml.gz 5lo8_validation.cif.gz
5lo8_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/lo/5lo8
https://data.pdbj.org/pub/pdb/validation_reports/lo/5lo8 ftp://data.pdbj.org/pub/pdb/validation_reports/lo/5lo8
ftp://data.pdbj.org/pub/pdb/validation_reports/lo/5lo8 リンク
リンク 集合体
集合体
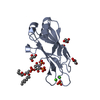
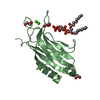
 要素
要素




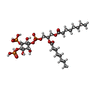





 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALBA
ALBA  / ビームライン: XALOC / 波長: 0.979 Å
/ ビームライン: XALOC / 波長: 0.979 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー








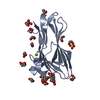







 PDBj
PDBj







