+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5klx | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal Structure of SMT Fusion Peptidyl-Prolyl Cis-Trans Isomerase from Burkholderia Pseudomallei Complexed with SF110 | ||||||
 要素 要素 | Chimera protein of Ubiquitin-like protein SMT3 and Peptidyl-prolyl cis-trans isomerase | ||||||
 キーワード キーワード | ISOMERASE / protein binding / BURKHOLDERIA / PEPTIDYL / PROLINE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SUMO is conjugated to E1 (UBA2:SAE1) / SUMOylation of nuclear envelope proteins / SUMO is transferred from E1 to E2 (UBE2I, UBC9) / SUMO is proteolytically processed / SUMOylation of transcription factors / SUMOylation of transcription cofactors / Postmitotic nuclear pore complex (NPC) reformation / septin ring / SUMOylation of DNA damage response and repair proteins / Transcriptional and post-translational regulation of MITF-M expression and activity ...SUMO is conjugated to E1 (UBA2:SAE1) / SUMOylation of nuclear envelope proteins / SUMO is transferred from E1 to E2 (UBE2I, UBC9) / SUMO is proteolytically processed / SUMOylation of transcription factors / SUMOylation of transcription cofactors / Postmitotic nuclear pore complex (NPC) reformation / septin ring / SUMOylation of DNA damage response and repair proteins / Transcriptional and post-translational regulation of MITF-M expression and activity / SUMOylation of DNA replication proteins / SUMOylation of SUMOylation proteins / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / SUMOylation of RNA binding proteins / SUMOylation of chromatin organization proteins / ubiquitin-like protein ligase binding / protein sumoylation / condensed nuclear chromosome / peptidylprolyl isomerase / peptidyl-prolyl cis-trans isomerase activity / protein tag activity / protein folding / metal ion binding / identical protein binding / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |   Burkholderia pseudomallei (類鼻疽菌) Burkholderia pseudomallei (類鼻疽菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.45 Å 分子置換 / 解像度: 2.45 Å | ||||||
 データ登録者 データ登録者 | Fox III, D. / Edwards, T.E. | ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Crystal Structure of SMT Fusion Peptidyl-Prolyl Cis-Trans Isomerase from Burkholderia Pseudomallei Complexed with SF110 著者: Lorimer, D.D. / Fox III, D. / Seufert, F. / Edwards, T.E. / Holzgrabe, U. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5klx.cif.gz 5klx.cif.gz | 288.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5klx.ent.gz pdb5klx.ent.gz | 230.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5klx.json.gz 5klx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5klx_validation.pdf.gz 5klx_validation.pdf.gz | 1.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5klx_full_validation.pdf.gz 5klx_full_validation.pdf.gz | 1.8 MB | 表示 | |
| XML形式データ |  5klx_validation.xml.gz 5klx_validation.xml.gz | 30.9 KB | 表示 | |
| CIF形式データ |  5klx_validation.cif.gz 5klx_validation.cif.gz | 40.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kl/5klx https://data.pdbj.org/pub/pdb/validation_reports/kl/5klx ftp://data.pdbj.org/pub/pdb/validation_reports/kl/5klx ftp://data.pdbj.org/pub/pdb/validation_reports/kl/5klx | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3uf8S S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 4分子 ABCD
| #1: タンパク質 | 分子量: 23001.830 Da / 分子数: 4 / 断片: UNP Q12306 residues 13-98,UNP Q3JK38 residues 2-113 / 由来タイプ: 組換発現 由来: (組換発現)   Burkholderia pseudomallei (strain 1710b) (類鼻疽菌) Burkholderia pseudomallei (strain 1710b) (類鼻疽菌)株: ATCC 204508 / S288c, 1710b / 遺伝子: SMT3, YDR510W, D9719.15, BURPS1710b_A0907 / プラスミド: PET28-HISSMT / 発現宿主:  参照: UniProt: Q12306, UniProt: Q3JK38, peptidylprolyl isomerase |
|---|
-非ポリマー , 5種, 148分子 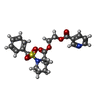








| #2: 化合物 | ChemComp-6UO / #3: 化合物 | #4: 化合物 | #5: 化合物 | ChemComp-PEG / | #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.17 Å3/Da / 溶媒含有率: 43.2 % |
|---|---|
| 結晶化 | 温度: 289 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 6.5 詳細: BUPSA.00130.A.D21 (CID4597, SMT TAG ON, BATCH 1264062) AT 10.37MG/ML (IN 25MM TRIS, PH8.0, 200MM NACL, 1% GLYCEROL, 1MM TCEP BUFFER) WAS INCUBATED WITH 2MM SF110_S (BSI5665). CRYSTALS WERE ...詳細: BUPSA.00130.A.D21 (CID4597, SMT TAG ON, BATCH 1264062) AT 10.37MG/ML (IN 25MM TRIS, PH8.0, 200MM NACL, 1% GLYCEROL, 1MM TCEP BUFFER) WAS INCUBATED WITH 2MM SF110_S (BSI5665). CRYSTALS WERE PRODUCED BY SITTING DROP VAPOR IFFUSION WITH AN EQUAL VOLUME COMBINATION OF THE PROTEIN/LIGAND COMPLEX AND A SOLUTION CONTAINING 10% W/V PEG20,000, 20% V/V PEG MME 550, 0.03M MGCL2, 0.03M CACL2, 0.1M MES/IMIDAZOLE, PH6.5 (MORPHEUS A1). CRYSTAL TRACKING ID 273103A1, RVA0-10, VAPOR DIFFUSION, SITTING DROP, TEMPERATURE 289K PH範囲: 6.5 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 21-ID-G / 波長: 0.97857 Å / ビームライン: 21-ID-G / 波長: 0.97857 Å |
| 検出器 | タイプ: MARMOSAIC 300 mm CCD / 検出器: CCD / 日付: 2016年6月3日 / 詳細: BERYLLIUM LENSES |
| 放射 | モノクロメーター: DIAMOND [111] / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.97857 Å / 相対比: 1 |
| 反射 | 解像度: 2.45→50 Å / Num. obs: 29732 / % possible obs: 99.4 % / Observed criterion σ(I): -3 / 冗長度: 3.7 % / Biso Wilson estimate: 47.15 Å2 / Rmerge(I) obs: 0.075 / Net I/σ(I): 14.99 |
| 反射 シェル | 解像度: 2.45→2.51 Å / 冗長度: 3.8 % / Rmerge(I) obs: 0.569 / Mean I/σ(I) obs: 2.26 / % possible all: 99.7 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3uf8 解像度: 2.45→37.79 Å / SU ML: 0.3 / 交差検証法: FREE R-VALUE / 位相誤差: 28.11
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 56.54 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.45→37.79 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.45→2.53 Å / Total num. of bins used: 11
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj













