| 登録情報 | データベース: PDB / ID: 5jk8
|
|---|
| タイトル | Phenylalanine hydroxylase from dictyostelium - BH2, norleucine complex |
|---|
 要素 要素 | Phenylalanine-4-hydroxylase |
|---|
 キーワード キーワード | OXIDOREDUCTASE / Phenylalanine hydroxylase / Dictyostelium / enzyme regulation / tetrahydrobiopterin |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Catecholamine biosynthesis / Serotonin and melatonin biosynthesis / Phenylalanine metabolism / prephenate dehydratase activity / tryptophan 5-monooxygenase / tryptophan 5-monooxygenase activity / asexual reproduction / phenylalanine 4-monooxygenase / phenylalanine 4-monooxygenase activity / tyrosine biosynthetic process ...Catecholamine biosynthesis / Serotonin and melatonin biosynthesis / Phenylalanine metabolism / prephenate dehydratase activity / tryptophan 5-monooxygenase / tryptophan 5-monooxygenase activity / asexual reproduction / phenylalanine 4-monooxygenase / phenylalanine 4-monooxygenase activity / tyrosine biosynthetic process / L-phenylalanine biosynthetic process / L-phenylalanine catabolic process / tetrahydrobiopterin binding / iron ion binding / identical protein binding類似検索 - 分子機能 Prephenate dehydratase, conserved site / Eukaryotic phenylalanine-4-hydroxylase, catalytic domain / Phenylalanine Hydroxylase / Aromatic amino acid hydroxylase / Tyrosine 3-monooxygenase-like / Aromatic amino acid hydroxylase, iron/copper binding site / Biopterin-dependent aromatic amino acid hydroxylases signature. / Aromatic amino acid hydroxylase / Aromatic amino acid hydroxylase, C-terminal / Aromatic amino acid monoxygenase, C-terminal domain superfamily ...Prephenate dehydratase, conserved site / Eukaryotic phenylalanine-4-hydroxylase, catalytic domain / Phenylalanine Hydroxylase / Aromatic amino acid hydroxylase / Tyrosine 3-monooxygenase-like / Aromatic amino acid hydroxylase, iron/copper binding site / Biopterin-dependent aromatic amino acid hydroxylases signature. / Aromatic amino acid hydroxylase / Aromatic amino acid hydroxylase, C-terminal / Aromatic amino acid monoxygenase, C-terminal domain superfamily / Aromatic amino acid hydroxylase superfamily / Biopterin-dependent aromatic amino acid hydroxylase / Biopterin-dependent aromatic amino acid hydroxylase family profile. / ACT domain profile. / ACT domain / ACT-like domain / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 : / 7,8-DIHYDROBIOPTERIN / NORLEUCINE / Phenylalanine-4-hydroxylase類似検索 - 構成要素 |
|---|
| 生物種 |   Dictyostelium discoideum (キイロタマホコリカビ) Dictyostelium discoideum (キイロタマホコリカビ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.394 Å シンクロトロン / 解像度: 2.394 Å |
|---|
 データ登録者 データ登録者 | Zhuang, N. / Lee, K.H. |
|---|
| 資金援助 |  韓国, 1件 韓国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National research foundation | |  韓国 韓国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Phenylalanine hydroxylase from dictyostelium - BH2, norleucine complex
著者: Zhuang, N. / Lee, K.H. |
|---|
| 履歴 | | 登録 | 2016年4月26日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2017年5月3日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年11月20日 | Group: Advisory / Data collection ...Advisory / Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_unobs_or_zero_occ_atoms / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.394 Å
シンクロトロン / 解像度: 2.394 Å  データ登録者
データ登録者 韓国, 1件
韓国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5jk8.cif.gz
5jk8.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5jk8.ent.gz
pdb5jk8.ent.gz PDB形式
PDB形式 5jk8.json.gz
5jk8.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5jk8_validation.pdf.gz
5jk8_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5jk8_full_validation.pdf.gz
5jk8_full_validation.pdf.gz 5jk8_validation.xml.gz
5jk8_validation.xml.gz 5jk8_validation.cif.gz
5jk8_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/jk/5jk8
https://data.pdbj.org/pub/pdb/validation_reports/jk/5jk8 ftp://data.pdbj.org/pub/pdb/validation_reports/jk/5jk8
ftp://data.pdbj.org/pub/pdb/validation_reports/jk/5jk8 リンク
リンク 集合体
集合体
 要素
要素

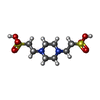
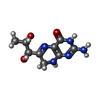







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト: PAL/PLS
シンクロトロン / サイト: PAL/PLS  / ビームライン: 6C1 / 波長: 1.2399 Å
/ ビームライン: 6C1 / 波長: 1.2399 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj







