| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| PHENIX | (1.10_2155: ???)| 精密化 | | xia2 | | データ削減 | | xia2 | | データスケーリング | | PHASER | | 位相決定 | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 3→47.227 Å / SU ML: 0.38 / 交差検証法: FREE R-VALUE / σ(F): 1.38 / 位相誤差: 24.23 / 立体化学のターゲット値: ML 分子置換 / 解像度: 3→47.227 Å / SU ML: 0.38 / 交差検証法: FREE R-VALUE / σ(F): 1.38 / 位相誤差: 24.23 / 立体化学のターゲット値: ML
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.3071 | 251 | 4.34 % |
|---|
| Rwork | 0.2496 | - | - |
|---|
| obs | 0.2522 | 5784 | 98.65 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL |
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 3→47.227 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 1416 | 0 | 0 | 0 | 1416 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.011 | 1429 | | X-RAY DIFFRACTION | f_angle_d| 1.446 | 1941 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 17.59 | 867 | | X-RAY DIFFRACTION | f_chiral_restr| 0.089 | 228 | | X-RAY DIFFRACTION | f_plane_restr| 0.008 | 267 | | | | | |
|
|---|
| LS精密化 シェル | | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Refine-ID | % reflection obs (%) |
|---|
| 2.9937-3.7716 | 0.3578 | 127 | 0.2675 | 2675 | X-RAY DIFFRACTION | 99 | | 3.7716-47.2332 | 0.2833 | 124 | 0.2407 | 2858 | X-RAY DIFFRACTION | 98 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 2.155 | -1.8639 | 1.3961 | 2.3702 | -4.0238 | 2 | 0.0369 | 0.728 | -0.4272 | -0.7865 | -0.2551 | -0.0151 | 0.2358 | 0.1716 | 0.2275 | 1.757 | -0.3102 | 0.3699 | 1.7087 | -0.5441 | 0.8544 | 12.7255 | -24.1953 | 2.9894 | | 2 | 1.6023 | 0.6796 | -0.3532 | 0.8822 | -0.3603 | 0.1524 | -0.2842 | 0.6405 | 0.2357 | -0.0194 | 0.0888 | -0.4474 | -0.0938 | 0.252 | -0.0429 | 0.6051 | 0.0512 | -0.119 | 2.235 | -0.2591 | 0.9143 | 8.4759 | -16.7201 | 6.2026 | | 3 | 1.1025 | 0.5769 | 0.9299 | 1.1687 | 0.3686 | 3.052 | -0.0013 | 0.1079 | -0.2122 | 0.3694 | 0.1967 | -0.2471 | 1.2595 | 0.0945 | 0.0129 | 0.7964 | -0.0515 | 0.0027 | 1.6323 | -0.5084 | 0.6552 | 8.6015 | -23.486 | 14.5815 | | 4 | 0.8964 | 0.1154 | -0.3332 | 1.6939 | 1.1901 | 1.9133 | -0.1098 | -0.0797 | -0.0357 | 0.1481 | 0.3471 | -0.2371 | 0.1215 | 0.3476 | -0.0153 | 0.3576 | 0.0882 | -0.0123 | 1.424 | -0.243 | 0.4065 | 13.0846 | -16.9214 | 18.0541 | | 5 | 1.5281 | -1.1278 | 1.3371 | 4.3076 | -2.7804 | 5.7471 | 0.0284 | -0.3726 | 0.0085 | -0.1554 | 0.1438 | 0.0463 | 0.5826 | -0.2293 | -0.1401 | 0.5481 | -0.2877 | 0.0878 | 1.3368 | -0.2875 | 0.4034 | 2.3434 | -22.9599 | 21.5031 | | 6 | 4.7906 | -0.6335 | -2.4808 | 3.588 | 0.2061 | 2.4242 | -0.1885 | -0.0002 | -0.2136 | -0.1148 | 0.0748 | -0.3761 | 0.2261 | 0.287 | -0.0453 | 0.4564 | 0.0841 | 0.0923 | 1.317 | -0.3865 | 0.6042 | 12.358 | -16.2854 | 22.4938 | | 7 | 0.5094 | 0.7081 | 1.237 | 4.3987 | 0.1567 | 3.7176 | 0.3625 | -0.3687 | -0.0884 | -0.0464 | -0.0777 | 0.1599 | 0.7068 | -0.2376 | -0.3498 | 0.6782 | -0.1545 | -0.1373 | 1.1214 | -0.3064 | 0.4786 | 1.9851 | -21.4753 | 25.9964 | | 8 | 7.2057 | 1.6406 | -1.1494 | 1.4017 | -1.2318 | 3.0854 | -0.3043 | 0.5445 | -1.0947 | -0.0488 | 0.1381 | -0.062 | 0.3686 | 0.0238 | 0.1702 | 0.325 | -0.0541 | 0.0752 | 1.0668 | -0.4074 | 0.5715 | 10.6839 | -18.3935 | 29.7208 | | 9 | 2.0376 | 0.7616 | -1.2379 | 0.285 | -0.4728 | 1.0251 | 0.053 | 0.1569 | 0.5318 | 0.0823 | -0.0526 | 0.3896 | 0.1196 | -0.2675 | 0.0473 | 0.3246 | -0.2369 | 0.0287 | 0.9422 | -0.1465 | 0.5181 | 4.8148 | -11.7375 | 32.7623 | | 10 | 2.9481 | 0.3296 | -0.1737 | 1.0921 | -0.165 | 1.6121 | -0.0259 | 0.5567 | -0.2955 | -0.0607 | -0.0814 | 0.0376 | 0.1513 | -0.094 | -0.0056 | 0.0728 | -0.1197 | -0.0362 | 0.2878 | -0.0177 | 0.4093 | 8.4073 | -12.8155 | 44.4758 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details |
|---|
| 1 | X-RAY DIFFRACTION | 1 | (chain A and resid 24:28)| 2 | X-RAY DIFFRACTION | 2 | (chain A and resid 29:41)| 3 | X-RAY DIFFRACTION | 3 | (chain A and resid 42:66)| 4 | X-RAY DIFFRACTION | 4 | (chain A and resid 67:78)| 5 | X-RAY DIFFRACTION | 5 | (chain A and resid 79:85)| 6 | X-RAY DIFFRACTION | 6 | (chain A and resid 86:98)| 7 | X-RAY DIFFRACTION | 7 | (chain A and resid 99:105)| 8 | X-RAY DIFFRACTION | 8 | (chain A and resid 106:133)| 9 | X-RAY DIFFRACTION | 9 | (chain A and resid 134:143)| 10 | X-RAY DIFFRACTION | 10 | (chain A and resid 144:213) | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3 Å
分子置換 / 解像度: 3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5dra.cif.gz
5dra.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5dra.ent.gz
pdb5dra.ent.gz PDB形式
PDB形式 5dra.json.gz
5dra.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5dra_validation.pdf.gz
5dra_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5dra_full_validation.pdf.gz
5dra_full_validation.pdf.gz 5dra_validation.xml.gz
5dra_validation.xml.gz 5dra_validation.cif.gz
5dra_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/dr/5dra
https://data.pdbj.org/pub/pdb/validation_reports/dr/5dra ftp://data.pdbj.org/pub/pdb/validation_reports/dr/5dra
ftp://data.pdbj.org/pub/pdb/validation_reports/dr/5dra リンク
リンク 集合体
集合体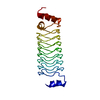

 要素
要素
 X線回折
X線回折 試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I04-1 / 波長: 0.92 Å
/ ビームライン: I04-1 / 波長: 0.92 Å 解析
解析 分子置換 / 解像度: 3→47.227 Å / SU ML: 0.38 / 交差検証法: FREE R-VALUE / σ(F): 1.38 / 位相誤差: 24.23 / 立体化学のターゲット値: ML
分子置換 / 解像度: 3→47.227 Å / SU ML: 0.38 / 交差検証法: FREE R-VALUE / σ(F): 1.38 / 位相誤差: 24.23 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj
