[English] 日本語
 Yorodumi
Yorodumi- PDB-5c5p: CRYSTAL STRUCTURE OF HUMAN TANKYRASE-2 IN COMPLEX WITH A PYRANOPY... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5c5p | ||||||
|---|---|---|---|---|---|---|---|
| Title | CRYSTAL STRUCTURE OF HUMAN TANKYRASE-2 IN COMPLEX WITH A PYRANOPYRIDONE INHIBITOR | ||||||
 Components Components | (Tankyrase-2) x 2 | ||||||
 Keywords Keywords | TRANSFERASE/TRANSFERASE INHIBITOR / WNT-SIGNALLING / BETA-CATENIN / PARP-DOMAIN / ADP-RIBOSYLATION / AXIN / TRANSFERASE-TRANSFERASE INHIBITOR COMPLEX | ||||||
| Function / homology |  Function and homology information Function and homology informationXAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity ...XAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / negative regulation of telomere maintenance via telomere lengthening / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity / pericentriolar material / Transferases; Glycosyltransferases; Pentosyltransferases / NAD+ poly-ADP-ribosyltransferase activity / positive regulation of telomere maintenance via telomerase / nucleotidyltransferase activity / TCF dependent signaling in response to WNT / Degradation of AXIN / Wnt signaling pathway / Regulation of PTEN stability and activity / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway / nuclear envelope / chromosome, telomeric region / Ub-specific processing proteases / Golgi membrane / perinuclear region of cytoplasm / enzyme binding / metal ion binding / nucleus / cytoplasm / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.75 Å MOLECULAR REPLACEMENT / Resolution: 1.75 Å | ||||||
 Authors Authors | Lukacs, C.M. / Janson, C.A. | ||||||
 Citation Citation |  Journal: Acs Med.Chem.Lett. / Year: 2015 Journal: Acs Med.Chem.Lett. / Year: 2015Title: Fragment-Based Drug Design of Novel Pyranopyridones as Cell Active and Orally Bioavailable Tankyrase Inhibitors. Authors: de Vicente, J. / Tivitmahaisoon, P. / Berry, P. / Bolin, D.R. / Carvajal, D. / He, W. / Huang, K.S. / Janson, C. / Liang, L. / Lukacs, C. / Petersen, A. / Qian, H. / Yi, L. / Zhuang, Y. / Hermann, J.C. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5c5p.cif.gz 5c5p.cif.gz | 111.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5c5p.ent.gz pdb5c5p.ent.gz | 83.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5c5p.json.gz 5c5p.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/c5/5c5p https://data.pdbj.org/pub/pdb/validation_reports/c5/5c5p ftp://data.pdbj.org/pub/pdb/validation_reports/c5/5c5p ftp://data.pdbj.org/pub/pdb/validation_reports/c5/5c5p | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5c5qC  5c5rC  3kr8S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 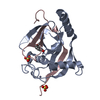
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Protein / Protein/peptide , 2 types, 4 molecules ABCD
| #1: Protein | Mass: 21824.545 Da / Num. of mol.: 2 / Fragment: UNP RESIDUES 946-1113 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: TNKS2, PARP5B, TANK2, TNKL / Production host: Homo sapiens (human) / Gene: TNKS2, PARP5B, TANK2, TNKL / Production host:  #2: Protein/peptide | Mass: 5493.216 Da / Num. of mol.: 2 / Fragment: UNP RESIDUES 1114-1162 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: TNKS2, PARP5B, TANK2, TNKL / Production host: Homo sapiens (human) / Gene: TNKS2, PARP5B, TANK2, TNKL / Production host:  |
|---|
-Non-polymers , 4 types, 454 molecules 






| #3: Chemical | | #4: Chemical | #5: Chemical | ChemComp-SO4 / #6: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.44 Å3/Da / Density % sol: 49.69 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, sitting drop / pH: 8.5 Details: 30-40% PEG 3350, 5% SATURATED AMMONIUM SULFATE, 0.1M TRIS PH 8.5 PH range: 8.5 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SLS SLS  / Beamline: X10SA / Wavelength: 0.9999 Å / Beamline: X10SA / Wavelength: 0.9999 Å |
| Detector | Type: PSI PILATUS 6M / Detector: PIXEL / Date: Oct 15, 2010 |
| Radiation | Monochromator: SI(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9999 Å / Relative weight: 1 |
| Reflection | Resolution: 1.75→45 Å / Num. obs: 53201 / % possible obs: 100 % / Observed criterion σ(I): 0 / Redundancy: 6.9 % / Biso Wilson estimate: 23.8 Å2 / Rmerge(I) obs: 0.045 / Net I/σ(I): 22.7 |
| Reflection shell | Resolution: 1.75→1.84 Å / Redundancy: 6.7 % / Rmerge(I) obs: 0.445 / Mean I/σ(I) obs: 4 / % possible all: 99.9 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 3KR8 Resolution: 1.75→45 Å / Rfactor Rfree error: 0.004 / Data cutoff high absF: 2154217.28 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / Cross valid method: THROUGHOUT / σ(F): 0
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: FLAT MODEL / Bsol: 58.91 Å2 / ksol: 0.4 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 27.9 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.75→45 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.75→1.86 Å / Rfactor Rfree error: 0.014 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 Movie
Movie Controller
Controller






















 PDBj
PDBj






