| 登録情報 | データベース: PDB / ID: 4xzh
|
|---|
| タイトル | Crystal structure of human Aldose Reductase complexed with NADP+ and JF0048 |
|---|
 要素 要素 | Aldose reductase |
|---|
 キーワード キーワード | OXIDOREDUCTASE / TIM barrel / Aldose reductase / diabetes / Halogenated compound / Cytosolic |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
glyceraldehyde oxidoreductase activity / Fructose biosynthesis / fructose biosynthetic process / L-glucuronate reductase activity / aldose reductase / D/L-glyceraldehyde reductase / glycerol dehydrogenase (NADP+) activity / C21-steroid hormone biosynthetic process / NADP-retinol dehydrogenase / Pregnenolone biosynthesis ...glyceraldehyde oxidoreductase activity / Fructose biosynthesis / fructose biosynthetic process / L-glucuronate reductase activity / aldose reductase / D/L-glyceraldehyde reductase / glycerol dehydrogenase (NADP+) activity / C21-steroid hormone biosynthetic process / NADP-retinol dehydrogenase / Pregnenolone biosynthesis / allyl-alcohol dehydrogenase / allyl-alcohol dehydrogenase activity / prostaglandin H2 endoperoxidase reductase activity / regulation of urine volume / metanephric collecting duct development / all-trans-retinol dehydrogenase (NADP+) activity / daunorubicin metabolic process / doxorubicin metabolic process / retinal dehydrogenase (NAD+) activity / aldose reductase (NADPH) activity / epithelial cell maturation / cellular hyperosmotic salinity response / retinoid metabolic process / renal water homeostasis / carbohydrate metabolic process / electron transfer activity / negative regulation of apoptotic process / mitochondrion / extracellular space / extracellular exosome / nucleoplasm / cytosol類似検索 - 分子機能 Aldo/keto reductase family putative active site signature. / Aldo/keto reductase family signature 1. / NADP-dependent oxidoreductase domain / Aldo/keto reductase family signature 2. / Aldo/keto reductase, conserved site / Aldo-keto reductase / NADP-dependent oxidoreductase domain / Aldo/keto reductase family / NADP-dependent oxidoreductase domain superfamily / TIM Barrel ...Aldo/keto reductase family putative active site signature. / Aldo/keto reductase family signature 1. / NADP-dependent oxidoreductase domain / Aldo/keto reductase family signature 2. / Aldo/keto reductase, conserved site / Aldo-keto reductase / NADP-dependent oxidoreductase domain / Aldo/keto reductase family / NADP-dependent oxidoreductase domain superfamily / TIM Barrel / Alpha-Beta Barrel / Alpha Beta類似検索 - ドメイン・相同性 Chem-48I / NADP NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE / Aldo-keto reductase family 1 member B1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1 Å 分子置換 / 解像度: 1 Å |
|---|
 データ登録者 データ登録者 | Cousido-Siah, A. / Ruiz, F.X. / Mitschler, A. / Dominguez, M. / de Lera, A.R. / Farres, J. / Pares, X. / Podjarny, A. |
|---|
 引用 引用 |  ジャーナル: Chemmedchem / 年: 2015 ジャーナル: Chemmedchem / 年: 2015
タイトル: Structural Determinants of the Selectivity of 3-Benzyluracil-1-acetic Acids toward Human Enzymes Aldose Reductase and AKR1B10.
著者: Ruiz, F.X. / Cousido-Siah, A. / Porte, S. / Dominguez, M. / Crespo, I. / Rechlin, C. / Mitschler, A. / de Lera, A.R. / Martin, M.J. / de la Fuente, J.A. / Klebe, G. / Pares, X. / Farres, J. / Podjarny, A. |
|---|
| 履歴 | | 登録 | 2015年2月4日 | 登録サイト: RCSB / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2015年11月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2016年1月27日 | Group: Database references |
|---|
| 改定 1.2 | 2024年1月10日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1 Å
分子置換 / 解像度: 1 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Chemmedchem / 年: 2015
ジャーナル: Chemmedchem / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4xzh.cif.gz
4xzh.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4xzh.ent.gz
pdb4xzh.ent.gz PDB形式
PDB形式 4xzh.json.gz
4xzh.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4xzh_validation.pdf.gz
4xzh_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4xzh_full_validation.pdf.gz
4xzh_full_validation.pdf.gz 4xzh_validation.xml.gz
4xzh_validation.xml.gz 4xzh_validation.cif.gz
4xzh_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xz/4xzh
https://data.pdbj.org/pub/pdb/validation_reports/xz/4xzh ftp://data.pdbj.org/pub/pdb/validation_reports/xz/4xzh
ftp://data.pdbj.org/pub/pdb/validation_reports/xz/4xzh リンク
リンク 集合体
集合体

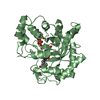
 要素
要素 Homo sapiens (ヒト) / 遺伝子: AKR1B1, ALDR1 / プラスミド: pET15b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: AKR1B1, ALDR1 / プラスミド: pET15b / 発現宿主: 
 X線回折
X線回折 試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06SA / 波長: 0.7085 Å
/ ビームライン: X06SA / 波長: 0.7085 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj