+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4xvd | ||||||
|---|---|---|---|---|---|---|---|
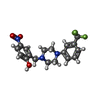
| タイトル | 17beta-HSD5 in complex with 4-nitro-2-({4-[3-(trifluoromethyl)phenyl]piperazin-1-yl}methyl)phenol | ||||||
 要素 要素 | Aldo-keto reductase family 1 member C3 | ||||||
 キーワード キーワード | OXIDOREDUCTASE/OXIDOREDUCTASE INHIBITOR / aldo-keto reductase inhibitor / OXIDOREDUCTASE-OXIDOREDUCTASE INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報prostaglandin-F synthase / testosterone 17beta-dehydrogenase (NADP+) / prostaglandin D2 11-ketoreductase activity / ketoreductase activity / prostaglandin F synthase activity / cellular response to prostaglandin stimulus / cellular response to corticosteroid stimulus / macromolecule metabolic process / 15-hydroxyprostaglandin-D dehydrogenase (NADP+) activity / 3beta(or 20alpha)-hydroxysteroid dehydrogenase ...prostaglandin-F synthase / testosterone 17beta-dehydrogenase (NADP+) / prostaglandin D2 11-ketoreductase activity / ketoreductase activity / prostaglandin F synthase activity / cellular response to prostaglandin stimulus / cellular response to corticosteroid stimulus / macromolecule metabolic process / 15-hydroxyprostaglandin-D dehydrogenase (NADP+) activity / 3beta(or 20alpha)-hydroxysteroid dehydrogenase / negative regulation of retinoic acid biosynthetic process / 5-alpha-androstane-3-beta,17-beta-diol dehydrogenase (NADP+) activity / Delta4-3-oxosteroid 5beta-reductase activity / farnesol catabolic process / geranylgeranyl reductase activity / 3alpha-hydroxysteroid 3-dehydrogenase / cellular response to jasmonic acid stimulus / 3alpha(17beta)-hydroxysteroid dehydrogenase (NAD+) / prostanoid biosynthetic process / testosterone dehydrogenase (NADP+) activity / androsterone dehydrogenase [NAD(P)+] activity / ketosteroid monooxygenase activity / regulation of testosterone biosynthetic process / RA biosynthesis pathway / testosterone biosynthetic process / : / 3alpha(or 20beta)-hydroxysteroid dehydrogenase / androstan-3-alpha,17-beta-diol dehydrogenase (NAD+) activity / Synthesis of bile acids and bile salts via 24-hydroxycholesterol / testosterone dehydrogenase (NAD+) activity / regulation of retinoic acid receptor signaling pathway / cellular response to prostaglandin D stimulus / progesterone metabolic process / 17beta-estradiol 17-dehydrogenase / retinal metabolic process / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / estradiol 17-beta-dehydrogenase [NAD(P)+] activity / all-trans-retinol dehydrogenase (NAD+) activity / : / prostaglandin H2 endoperoxidase reductase activity / all-trans-retinol dehydrogenase (NADP+) activity / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; NAD又はNADPを用いる / bile acid binding / daunorubicin metabolic process / doxorubicin metabolic process / retinal dehydrogenase (NAD+) activity / aldose reductase (NADPH) activity / oxidoreductase activity, acting on NAD(P)H, quinone or similar compound as acceptor / prostaglandin metabolic process / renal absorption / steroid metabolic process / positive regulation of endothelial cell apoptotic process / Synthesis of bile acids and bile salts via 27-hydroxycholesterol / Synthesis of bile acids and bile salts via 7alpha-hydroxycholesterol / retinoid metabolic process / Retinoid metabolism and transport / keratinocyte differentiation / response to nutrient / cellular response to calcium ion / cellular response to starvation / male gonad development / positive regulation of reactive oxygen species metabolic process / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / G protein-coupled receptor signaling pathway / positive regulation of cell population proliferation / extracellular exosome / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.81 Å 分子置換 / 解像度: 2.81 Å | ||||||
 データ登録者 データ登録者 | Amano, Y. / Yamaguchi, T. / Niimi, T. / Sakashita, H. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2015 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2015タイトル: Structures of complexes of type 5 17 beta-hydroxysteroid dehydrogenase with structurally diverse inhibitors: insights into the conformational changes upon inhibitor binding. 著者: Amano, Y. / Yamaguchi, T. / Niimi, T. / Sakashita, H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4xvd.cif.gz 4xvd.cif.gz | 139.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4xvd.ent.gz pdb4xvd.ent.gz | 109.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4xvd.json.gz 4xvd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4xvd_validation.pdf.gz 4xvd_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4xvd_full_validation.pdf.gz 4xvd_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  4xvd_validation.xml.gz 4xvd_validation.xml.gz | 26.3 KB | 表示 | |
| CIF形式データ |  4xvd_validation.cif.gz 4xvd_validation.cif.gz | 34.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xv/4xvd https://data.pdbj.org/pub/pdb/validation_reports/xv/4xvd ftp://data.pdbj.org/pub/pdb/validation_reports/xv/4xvd ftp://data.pdbj.org/pub/pdb/validation_reports/xv/4xvd | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: 1 / Refine code: 1
NCSアンサンブル:
NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 37977.371 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: AKR1C3 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: AKR1C3 / 発現宿主:  参照: UniProt: P42330, testosterone 17beta-dehydrogenase (NADP+) #2: 化合物 | #3: 化合物 | #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.6 Å3/Da / 溶媒含有率: 52.67 % |
|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 5.5 詳細: 0.1 M sodium citrate, 0.4 M ammonium acetate, 2.5% (v/v) 2-methyl-2,4-pentanediol, 22-30% (w/v) PEG4000 |
-データ収集
| 回折 | 平均測定温度: 90 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Photon Factory Photon Factory  / ビームライン: AR-NW12A / 波長: 1 Å / ビームライン: AR-NW12A / 波長: 1 Å |
| 検出器 | タイプ: ADSC QUANTUM 210 / 検出器: CCD / 日付: 2005年12月8日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→50 Å / Num. obs: 17267 / % possible obs: 90.4 % / 冗長度: 3.2 % / Net I/σ(I): 7.7 |
| 反射 シェル | 解像度: 2.8→2.9 Å / Rmerge(I) obs: 0.454 / Mean I/σ(I) obs: 2.6 / Num. unique obs: 1202 / % possible all: 91.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1S1P 解像度: 2.81→40.67 Å / Cor.coef. Fo:Fc: 0.91 / Cor.coef. Fo:Fc free: 0.854 / SU B: 17.076 / SU ML: 0.331 / 交差検証法: THROUGHOUT / ESU R Free: 0.431 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 33.592 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.81→40.67 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj