| 登録情報 | データベース: PDB / ID: 4ur0
|
|---|
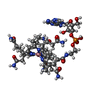
| タイトル | Crystal structure of the PCE reductive dehalogenase from S. multivorans in complex with trichloroethene |
|---|
 要素 要素 | TETRACHLOROETHENE REDUCTIVE DEHALOGENASE CATALYTIC SUBUNIT PCEA |
|---|
 キーワード キーワード | OXIDOREDUCTASE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
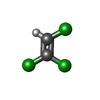
4 iron, 4 sulfur cluster binding / metal ion binding / identical protein binding類似検索 - 分子機能 Reductive dehalogenase domain / Reductive dehalogenase / Reductive dehalogenase subunit / Twin arginine translocation (Tat) signal profile. / Twin-arginine translocation pathway, signal sequence / 4Fe-4S ferredoxin, iron-sulphur binding, conserved site / 4Fe-4S ferredoxin-type iron-sulfur binding region signature. / 4Fe-4S ferredoxin-type iron-sulfur binding domain profile. / 4Fe-4S ferredoxin-type, iron-sulphur binding domain類似検索 - ドメイン・相同性 BENZAMIDINE / NORPSEUDO-B12 / IRON/SULFUR CLUSTER / 1,1,2-trichloroethene / Tetrachloroethene reductive dehalogenase catalytic subunit PceA類似検索 - 構成要素 |
|---|
| 生物種 |  SULFUROSPIRILLUM MULTIVORANS (バクテリア) SULFUROSPIRILLUM MULTIVORANS (バクテリア) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 1.798 Å 単波長異常分散 / 解像度: 1.798 Å |
|---|
 データ登録者 データ登録者 | Bommer, M. / Kunze, C. / Fesseler, J. / Schubert, T. / Diekert, G. / Dobbek, H. |
|---|
 引用 引用 |  ジャーナル: Science / 年: 2014 ジャーナル: Science / 年: 2014
タイトル: Structural Basis for Organohalide Respiration.
著者: Bommer, M. / Kunze, C. / Fesseler, J. / Schubert, T. / Diekert, G. / Dobbek, H. |
|---|
| 履歴 | | 登録 | 2014年6月25日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2014年10月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年11月5日 | Group: Database references |
|---|
| 改定 1.2 | 2017年7月12日 | Group: Refinement description / カテゴリ: software / Item: _software.name |
|---|
| 改定 1.3 | 2019年3月6日 | Group: Data collection / Experimental preparation / カテゴリ: exptl_crystal_grow / Item: _exptl_crystal_grow.method |
|---|
| 改定 1.4 | 2024年5月8日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Other
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_sf / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 SULFUROSPIRILLUM MULTIVORANS (バクテリア)
SULFUROSPIRILLUM MULTIVORANS (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 1.798 Å
単波長異常分散 / 解像度: 1.798 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Science / 年: 2014
ジャーナル: Science / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4ur0.cif.gz
4ur0.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4ur0.ent.gz
pdb4ur0.ent.gz PDB形式
PDB形式 4ur0.json.gz
4ur0.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4ur0_validation.pdf.gz
4ur0_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4ur0_full_validation.pdf.gz
4ur0_full_validation.pdf.gz 4ur0_validation.xml.gz
4ur0_validation.xml.gz 4ur0_validation.cif.gz
4ur0_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ur/4ur0
https://data.pdbj.org/pub/pdb/validation_reports/ur/4ur0 ftp://data.pdbj.org/pub/pdb/validation_reports/ur/4ur0
ftp://data.pdbj.org/pub/pdb/validation_reports/ur/4ur0 リンク
リンク 集合体
集合体
 要素
要素 SULFUROSPIRILLUM MULTIVORANS (バクテリア)
SULFUROSPIRILLUM MULTIVORANS (バクテリア)








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  BESSY
BESSY  / ビームライン: 14.1 / 波長: 0.9184
/ ビームライン: 14.1 / 波長: 0.9184  解析
解析 単波長異常分散
単波長異常分散 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj












